生物資訊學中的資料管理/ER理論
| 章節導航 | |
| 頂部 | E/R理論 - 規範化 - 資料查詢 - 整合SQL和程式語言 |
生物體被歸類為原核生物和真核生物。分子生物學中心法則規定DNA被轉錄成mRNA,然後被翻譯成蛋白質。蛋白質在細胞中執行生命功能。
細胞中的遺傳物質由幾條DNA雙螺旋鏈組成,形成超螺旋結構。DNA分子由糖磷酸骨架組成,在其上核苷酸形成鹼基對。4個鹼基用字母A、C、T和G表示。在DNA雙螺旋中,T與A配對,C與G配對。
- 基因組
- GenBank:由NIH維護的,包含所有公開可用DNA序列的集合。 [1]
- 蛋白質
- PDB:蛋白質資料庫 [2]
- 功能和註釋
- 相互作用資料庫 - 蛋白質-蛋白質相互作用
- BIND:生物分子相互作用網路資料庫 [3]
- 通路
- 化學反應通路:KEGG京都基因和基因組百科全書
- 訊號轉導(例如,對外部訊號的反應):STKE訊號轉導知識環境 [4]
- 文字
- PubMed:生物學研究文章的集合。 [5]
- 生物體特定資料庫
《核酸研究》雜誌的1月期刊是生物資料庫目錄。
- 資料模型
- 關係型
- 面向物件
- 層次型
- 半結構化
- 非結構化(例如文字)
- 查詢模型:SQL
- 資訊整合
- 資料探勘
- 集合
- 物件的集合
- 設和是兩個集合,和
- 笛卡爾積
- 的是包含所有可能的對的集合,從集合中取一個物件,從集合中取另一個物件。
- A × B = {(a,1),(a,2),(a,4),(b,1),(b,2),(b,4),(c,1),(c,2),(c,4)}
- 關係
- 是笛卡爾積的子集。
| 基因ID | 名稱 | 註釋 | 實驗ID | 實驗描述 | 表達水平 |
|---|---|---|---|---|---|
| g2347 | p53 | 腫瘤抑制基因 | e0 | 熱休克 | +2 |
| g2347 | p53 | 腫瘤抑制基因 | e1 | 冷休克 | -1 |
| g2347 | p53 | 腫瘤抑制基因 | e2 | 乾燥 | 0 |
| ... | ... | ... | ... | ... | ... |
以上是一個非規範化關係的示例。它存在以下問題:
- 冗餘:基因資訊在不同的實驗中重複出現,並且實驗資訊可能在多個基因中出現。
- 更新異常:如果需要更新基因的註釋,則需要更新表的幾行。
- 刪除異常:刪除基因會導致丟失有關實驗的資訊。
上圖展示了 E/R 模型在資料庫設計中的應用。我們首先從想要建模的資訊入手,將其表達在 E/R 模型中。然後,我們將抽象的 E/R 設計轉換為某個 DBMS 的資料規範語言中的模式。最常見的是,DBMS 使用關係模型。如果是這樣,那麼透過一個相當機械化的過程,抽象設計將被轉換為一個具體的、關係型的設計,稱為“關係資料庫模式”。
| 實體(名詞) | 關係(動詞) |
|---|---|
| 基因 | 被用於 |
| 實驗 |
讀取雙向關係
[edit | edit source]
多對多關係(無箭頭)
- 一個 A **可以** 與多個 B 相關聯,例如一個 **基因** 可以 **被用於** 多個 **實驗**
- 一個 B **可以** 與多個 A 相關聯,例如一個 **實驗** 可以 **用於** 多個 **基因**

一對一(兩側都有箭頭)
- 一個 A **可以** 與 **最多** 一個 B 相關聯,例如一個 **DNA 分子** 可以 **轉錄** 為最多一個 **RNA 初級轉錄本**
- 一個 B **可以** 與 **最多** 一個 A 相關聯,例如一個 **RNA 初級轉錄本** 可以 **轉錄** 自最多一個 **DNA 分子**

多對一(一個箭頭指向 **一** 方)
- 一個 A **可以** 與 **最多** 一個 B 相關聯,例如一個 **密碼子** 可以 **編碼** 為最多一個 **氨基酸**
- 一個 B **可以** 與 **多個** A 相關聯,例如一個 **氨基酸** 可以 **編碼** 由多個 **密碼子**
三方關係
[edit | edit source]
S => 學生,C => 課程,I => 教師
- 任何 (S,C) 對 **可以** 擁有多個 I
- 任何 (S,I) 對 **可以** 擁有多個 C
- 任何 (C,I) 對 **可以** 擁有多個 S
替換三方關係
[edit | edit source]以下是以另一個三方關係(三元關係)為例。
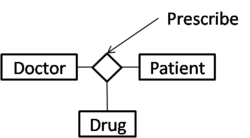
該關係可以用一組二元關係來代替,如下所示。
例如:
A => 醫生,B => 患者,C => 藥物,R => 開處方
| R | A | B | C |
|---|---|---|---|
| r1 | a1 | b1 | c2 |
| r2 | a1 | b2 | c1 |
| r3 | a2 | b1 | c1 |
| r4 | a2 | b1 | c2 |
然而,該圖與以下圖並不等效。

圖中的雙向關係為 (A,B)、(B,C) 和 (C,A)。這三個二元關係與給定的三元關係有著不同的含義。例如,一組醫生與特定患者和藥物對的關聯無法用二元關係來建模。
它是對是否應該將關係表示為三元關係還是一組二元關係(如上所述)進行建模的決策。
框定雙向關係
[edit | edit source]通常,框定某事物意味著該實體很重要,需要擁有自己的空間,而不是一個用來建模關係的概念。當框定關係時,實體的名稱通常會成為關係名稱中動詞的片語。
例如,以下 E/R 模型。

該模型的表示例如下:
| 出生 | 母親 | 嬰兒 |
|---|---|---|
| b1 | 蘇 | 湯姆 |
| b2 | 蘇 | 瑪麗 |
| b3 | 簡 | 羅伯特 |
以下模型也可以用來代替原始模型,但它與之前的模型有著不同的含義。該模型的表示例如下:
| 出生 | 母親 | 嬰兒 |
|---|---|---|
| b1 | 蘇 | 湯姆 |
| b1 | 蘇 | 瑪麗 |
| b2 | 簡 | 羅伯特 |
這種關係的框定提供了更大的靈活性。
屬性,鍵
[edit | edit source]實體或關係的屬性稱為屬性。屬性表示為附加在實體或關係上的氣球。屬性也需要作為列出現在表中。
基因表達過程可以分為幾個步驟,轉錄過程是其中一個步驟。mRNA 透過轉錄過程從 DNA 合成。同樣,cDNA 透過逆轉錄過程從 mRNA 合成。微陣列是一種晶片,在其表面排列著大量 DNA 序列。在 DNA 微陣列實驗中,合成的 cDNA 被倒入晶片,cDNA 透過雜交與晶片上的互補 DNA 序列結合。測量結合的 cDNA 量可以量化實驗中的基因表達水平。
如前所述,關係也可以有屬性。
以下是以具有屬性的關係為例。

一個屬性或一組屬性,其值對於每個實體例項都是唯一的,稱為候選鍵。一個實體可以有多個候選鍵,選定用來標識實體的候選鍵稱為(主)鍵。在 ER 圖中,鍵的名稱用下劃線標記。例如,ssn 和 sid 對於所有學生都是唯一的,因此兩者都是候選鍵。ssn 或 sid 可以被選作實體型別“學生”的主鍵。
從 ERD 建立表
[edit | edit source]將 E/R 設計轉換為關係資料庫模式是一個相當機械化和直接的過程。
- 將每個實體集轉換為具有相同屬性集的關係,以及
- 用一個關係來代替一個關係,該關係的屬性是連線的實體集的鍵
對於
- 多對多關係 - 表示關係的關係的鍵必須是參與實體集的鍵的並集
- 多對一關係 - 表示關係的關係的鍵必須來自多的一方
- 一對一關係 - 參與實體集的任何一方的鍵都可以作為關係的鍵
例如:

'takes' 的鍵取決於關係的基數。每個關係基數的鍵是
- 多對多關係 -> a,c
- 一對多關係 -> c
- 一對一關係 -> a 或 c
如果 'takes' 是一個一對多基數關係,則從模型生成的表將是
以下是 3 元關係生成表的示例。A、B 和 C 的表格被省略了。



以下兩個表格中其中一個可能是可能的。
以下是一個一元關係的示例。
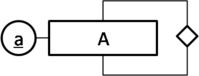
以下是一個其他示例。

在不同的州,可能存在許多同名的縣。例如,名為 Montgomery 的縣位於弗吉尼亞州、德克薩斯州、馬里蘭州等等。因此,縣的名字不能作為縣實體的鍵。然而,可以透過組合州名和縣名來獲取縣的鍵。這樣的實體在它們的屬性中沒有鍵被稱為弱集或弱實體。這樣的實體的鍵可以從某些相關實體的屬性中獲取。相關實體通常被稱為識別實體。從弱集到其識別實體的關係被稱為識別關係。

上面的 ER 圖顯示了州和縣之間的二元關係。雙邊框矩形表示弱集,識別實體是州,因為州的兩個字元和縣名的組合識別了一個縣。圖中雙邊框關係的符號也表示識別關係。識別實體可以透過搜尋雙邊框菱形並檢視從菱形發出的箭頭來找到。

弱集是實體集,它們不提供它們自己的所有鍵(參見圖)。它們依賴於其他實體集來提供一些或所有鍵。我們使用以下方式對 E/R 圖進行編碼
請注意, 中沒有寫兩次 。我們不需要完全編碼弱關係。它隱含於 的編碼中。

我們將下一個 E/R 圖的實體集編碼為
關於弱集的問題
[edit | edit source]
不,它無效。對於 的任何例項,必須只有一個 ,否則它將具有相同型別鍵的多個例項。應該有一條指向 的箭頭。

不,它無效。此圖表示
- 對於每個 ,,都有一個
- 對於每個 ,,都有許多 s
- 對於每個 ,,都有一個 }
但這並不排除一個 具有多個 s 的可能性。沒有明確的方法可以繪製一個弱集,該弱集從其他兩個實體集中提取鍵,而沒有使用 E/R 圖繪製兩個單獨的關係。
弱集的起因
[edit | edit source]
- 將關係轉換為實體集(參見圖)
- 層次結構
注意圖 1 中,所有原始實體集都有指向它們的箭頭。我們需要這些箭頭,因為每個關係實體只能擁有一個, 和 。
層次結構是一個有趣的情況,它引入了我們到繼承的概念。
在本節中,我們將討論繼承;什麼是繼承,為什麼要使用繼承,以及如何使用繼承。
繼承是指一個事物(一個實體集)是另一個事物(另一個實體集)的子集。
檢查一個實體集 是否從另一個實體集 繼承的一種方法是問:“ 是一個 ”。
繼承是指當你能坦然地說“$A$ 是一個 $B$”,而且當你競選公職時,沒有人會指出你說過這句話。~Naren Ramakrishnan
以下哪些是有效的繼承關係?
- 蘋果 是一個 水果
- 蘋果 是一個 牛
- g23 是一個 基因
- 第 1 個是正確的,因為蘋果是一種水果。第 2 個是不正確的,因為蘋果不是牛。然而,第 3 個也不正確,因為 g23 是一個特定的基因,它並不能完全體現我們想要在繼承中看到的概念。一個更好的測試繼承的方法是,如果你能說“$A$ 是一種 $B$”。
我們使用繼承是為了賦予實體集的子集其父實體集的屬性,並讓子集在此基礎上擴充套件額外的資訊。當我們說“$A$ 是一個 $B$”時,我們已經在傳達很多資訊。例如,當我們說“蘋果 是一個 水果”時,即使我們不太瞭解“蘋果”是什麼,我們也已經傳達了“蘋果”有種子,是更大植物的一部分,並且比典型的蔬菜更美味。

觀察這個圖。哺乳動物是動物,爬行動物也是。它們都有 id、屬、重量、種和安全等級(無論那意味著什麼)。哺乳動物和爬行動物都繼承了這些屬性。需要注意的是,在繼承關係中,子實體集繼承了父實體集的鍵。在這個例子中,哺乳動物和爬行動物的鍵都是它們的動物 id。
我們之所以要為哺乳動物和爬行動物建立單獨的實體集,是因為我們想為它們新增屬性。注意哺乳動物有毛髮數量,爬行動物有蛋的數量。我們不需要建立一個全新的實體集來解釋這一點,我們只需要使用繼承來說明哺乳動物是一種有毛髮數量的動物,而爬行動物是一種有蛋數量的動物。有些動物既有毛髮數量,也有蛋的數量。我們稱這些動物為“怪人”。從圖中可以看出,如果我們只使用多重繼承,讓“怪人”既是哺乳動物又是爬行動物,我們就不需要為“怪人”新增任何額外的屬性。隱含地,“怪人”也是動物,並且具有動物的所有屬性,包括鍵 id。換句話說,繼承是傳遞性的。
我們將圖中的實體集編碼為
我們不需要編碼“怪人”,它們是隱含的。注意,雖然弱集有不同的鍵,但繼承集沒有不同的鍵。還要注意,繼承集會獲得其父集的所有屬性,而不僅僅是一部分。

是的,這取決於關係。考慮農村縣和州之間的關係,關係是“出售給”。還要注意,由於我們具有單一繼承,因此我們必須為農村縣新增屬性。

是的,這可能有效。考慮新的關係也為“出售給”。




























