R 中的資料探勘演算法/聚類/圍繞中心點劃分 (PAM)
聚類是一種無監督機器學習演算法,它將資料集中的實體分組,這些實體在同一個簇中具有高度相似性。如今,許多領域都在使用這些型別的演算法以自動化的方式將資料集分成組,並且仍然可以獲得高質量的結果。
聚類過程不是一個通用的過程,因為存在許多組資料集,對於其中的一些資料集,使用的度量型別是相關的,而對於其他資料集,代表每個簇的實體更有趣。與資料集組類似,存在許多聚類演算法,每個演算法都試圖利用資料型別,因此,它們中的每一個都更適合特定型別的資料。
本節將更詳細地解釋圍繞中心點劃分 (PAM) 演算法,展示演算法的工作原理、引數以及含義、資料集示例、如何執行演算法以及使用資料集作為輸入的執行結果。
PAM 演算法由 Leonard Kaufman 和 Peter J. Rousseeuw 開發,該演算法與 K 均值非常相似,主要是因為兩者都是劃分演算法,換句話說,兩者都將資料集分成組(簇),並且兩者都試圖最小化誤差,但 PAM 使用中心點,中心點是代表其插入的組的資料集中的實體,而 K 均值使用中心點,中心點是人工建立的代表其簇的實體。
PAM 演算法將包含 n 個物件的 資料集 分成 k 個簇,其中資料集和 k 都作為演算法的輸入。該演算法使用不相似矩陣,其目標是最小化每個簇的代表與其成員之間的總體不相似性。該演算法使用以下模型來解決問題:
受制於
其中 F(x) 是要最小化的主要函式,d(i,j) 是實體 i 和 j 之間的不相似性度量,而 zij 是一個確保僅計算來自同一簇的實體之間的不相似性的變數。其他表示式是約束條件,它們具有以下功能:(1.) 確保每個實體都分配給一個且僅一個簇,(2.) 確保實體分配給代表該簇的中心點,(3.) 確保恰好有 k 個簇,以及 (4.) 允許決策變數僅取 0 或 1 的值。
PAM 演算法可以處理兩種型別的輸入,第一個是代表每個實體及其變數值的矩陣,第二個是直接的不相似矩陣,在後一種情況下,使用者可以直接提供不相似性作為演算法的輸入,而不是代表實體的資料矩陣。無論哪種方式,該演算法都能夠找到問題的解決方案,在一般的分析中,該演算法按照以下方式進行
構建階段
- 1. 選擇 k 個實體作為中心點,或者如果這些實體已提供,則將它們用作中心點;
- 2. 如果未提供,則計算不相似矩陣;
- 3. 將每個實體分配到其最近的中心點;
交換階段
- 4. 對於每個簇,搜尋該簇中的任何實體是否可以降低平均不相似係數,如果可以,則選擇最能降低該係數的實體作為該簇的中心點;
- 5. 如果至少一箇中心點已更改,則轉到 (3),否則結束演算法。
這並沒有描述“PAM”演算法。這是一種使用中心點的 K 均值變體,而不是具有其特徵構建和交換階段的 PAM。
PAM 演算法的虛擬碼如下所示
演算法 1:PAM 演算法 輸入: E = {e1,e2,...en}(要聚類的 資料集 或不相似矩陣)
- k(簇的數量)
- 度量(在不相似矩陣上使用的度量型別)
- diss(指示 E 是否是不相似矩陣的標誌)
輸出: M = {m1,m2,...,mk}(簇中心點的向量)
- L = {l(e) | e = 1,2,...,n}(E 的簇標籤集)
foreach mi € M do
- mi ← ej € E;(例如,隨機選擇)
end if diss ≠ true
- 不相似性 ← 計算不相似矩陣(E, 度量);
else
- 不相似性 ← E;
end repeat
- foreach ei € E do
- l(ei) ← argminDissimilarity(ei, 不相似性, M);
- end
- 已更改 ← false;
- foreach mi € M do
- Mtmp ← 選擇最佳簇中心點(E, 不相似性, L);
- end
- if Mtmp ≠ M
- M ← Mtmp;
- 已更改 ← true;
- end
until 已更改 = true;
在 R 程式語言中,PAM 演算法在 cluster 包中可用,可以透過以下命令呼叫
pam(x, k, diss, metric, medoids, stand, cluster.only, do.swap, keep.diss, keep.data, trace.lev)
其中引數為
x:代表資料集實體的數值資料矩陣,或者可以是不相似矩陣,它取決於 diss 引數的值。如果 x 是資料矩陣,則每行都是一個實體,每列都是一個變數,在這種情況下,只要每對實體至少有一個不缺失的情況,就可以允許缺失值。如果 x 是不相似矩陣,則不允許缺失值。
k:資料集將被劃分的簇的數量,其中 0 < k < n,其中 n 是實體的數量。
diss:邏輯標誌,如果它是 TRUE,則 x 被用作不相似矩陣,如果它是 FALSE,則 x 將被視為資料矩陣。
度量:一個字串,指定將使用兩個度量標準中的每一個來計算不相似矩陣,度量變數可以是“歐幾里得”以使用歐幾里得距離,也可以是“曼哈頓”以使用曼哈頓距離。
stand:邏輯標誌,如果它是 TRUE,則在計算不相似性之前對 x 中的測量值進行標準化。透過減去列的平均值併除以變數的平均絕對偏差,對每列進行標準化測量。如果 x 是不相似矩陣,則忽略此引數。
cluster.only:邏輯標誌,如果它是 TRUE,則僅計算並返回聚類。
do.swap:邏輯標誌,指示是否應執行交換階段 (TRUE) 還是不執行 (FALSE)。
keep.diss: 邏輯標誌,指示是否在結果中保留差異(TRUE)或不保留(FALSE)。
keep.data: 邏輯標誌,指示是否在結果中保留輸入資料 x(TRUE)或不保留(FALSE)。
trace.lev: 一個數值引數,指定在演算法的構建和交換階段列印診斷資訊的跟蹤級別。預設值為 0,不列印任何內容。
PAM 演算法返回一個 pam 物件,其中包含有關演算法執行結果的資訊。
在 R 中,有兩種方法可以檢視 PAM 演算法的結果:第一種方法是列印演算法返回的物件,第二種方法是繪製物件中的資料,建立一個結果圖形。第一種視覺化資訊的方法理解起來稍微複雜一些,但它提供了更完整和準確的資訊,而第二種方法則容易理解得多,讓使用者能夠更好地檢視資訊並新增對他而言相關的其他資訊。
要以文字方式檢視 PAM 演算法執行結果的資料,有兩種方法:一種比較簡單,提供有關物件的更簡潔的資訊;另一種更詳細,提供更完整的資訊。在下面列出的兩個命令中,第一個命令以簡潔的方式列印資訊,而第二個命令則以更完整的方式列印資訊。
print (result) summary (result)
視覺化演算法執行結果資料的另一種方法是使用圖形,可以透過以下命令來實現。
plot (result)
示例:為了展示演算法的使用示例及其執行結果,使用了一個包含少量實體和維度的簡單資料集,如以下表格所示。
表 1:簡單資料集
| 物件 | 屬性 x | 屬性 y |
|---|---|---|
| 1 | 1 | 1 |
| 2 | 2 | 3 |
| 3 | 1 | 2 |
| 4 | 2 | 2 |
| 5 | 10 | 4 |
| 6 | 11 | 5 |
| 7 | 10 | 6 |
| 8 | 12 | 5 |
| 9 | 11 | 6 |
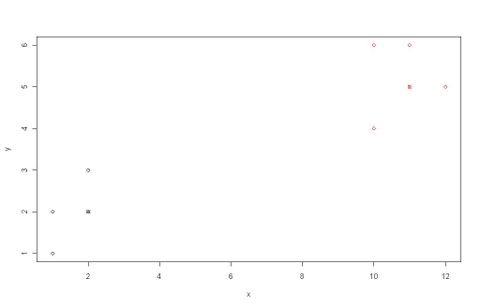
正如我們所見,資料被分成兩個聚類,因此我們將使用 k = 2。PAM 演算法可以按以下方式執行。
#load the table from a file x <- read.table(“table.txt”) #execute the pam algorithm with the dataset created for the example result <- pam(x, 2, FALSE, "euclidean") #print the results data in the screen summary(result) #plot a graphic showing the clusters and the medoids of each cluster plot(result$data, col = result$clustering) points(result$medoids, col = 1:2, pch = 4)
列印執行結果將得到
Medoids:
ID x y
4 4 2 2
6 6 11 5
Clustering vector:
1 2 3 4 5 6 7 8 9
1 1 1 1 2 2 2 2 2
Objective function:
build swap
1.255618 0.915849
Numerical information per cluster:
size max_diss av_diss diameter separation
[1,] 4 1.414214 0.8535534 2.236068 8.062258
[2,] 5 1.414214 0.9656854 2.236068 8.062258
Isolated clusters:
L-clusters: character(0)
L*-clusters: [1] 1 2
Silhouette plot information:
cluster neighbor sil_width
3 1 2 0.8898942
4 1 2 0.8788422
1 1 2 0.8549629
2 1 2 0.8297000
6 2 1 0.8790384
9 2 1 0.8631441
8 2 1 0.8425790
7 2 1 0.8232848
5 2 1 0.7747713
Average silhouette width per cluster:
[1] 0.8633498 0.8365635
Average silhouette width of total data set:
[1] 0.8484685
36 dissimilarities, summarized :
Min. 1st Qu. Median Mean 3rd Qu. Max.
1.0000 1.4142 8.3951 6.1559 9.9362 11.7050
Metric : euclidean
Number of objects : 9
Available components:
[1] "medoids" "id.med" "clustering" "objective" "isolation" "clusinfo"
"silinfo" "diss" "call"
[10] "data"
而繪製將得到
在本節中,我們將使用 PAM 來進行案例研究。
在本案例研究中,我們使用了 R 包 datasets 中提供的 iris 資料庫的一部分。這個著名的(費舍爾或安德森)虹膜資料集給出了 3 個虹膜種類的 50 朵花的萼片長度和寬度以及花瓣長度和寬度的測量值(單位為釐米)。這 3 個種類分別是山鳶尾(Iris setosa)、變色鳶尾(versicolor)和維吉尼亞鳶尾(virginica)。利用此資料集提供的資料,很自然地會想到驗證三種虹膜的每一種花的其他同類花是否真的相似。因此,在本案例研究中,我們將使用萼片和花瓣的長度和寬度將資料集聚類成 3 組,然後驗證這些聚類是否真的與花卉種類相匹配。
在本案例研究中使用的資料集包含以下列
- Flower: 花的 ID;
- Sepal.Length: 萼片長度的數值(單位為釐米);
- Sepal.Width: 萼片寬度的數值(單位為釐米);
- Petal.Length: 花瓣長度的數值(單位為釐米);
- Petal.Width: 花瓣寬度的數值(單位為釐米);
- Species: 用於識別花的種類。
輸入資料是一個表格,包含原始 iris 資料集的 50%(75 個實體),原始資料集包含 150 朵花,每個花有 5 個屬性。因此,在本案例研究中使用的資料集由以下表格表示。
表 2:從 iris 資料集中抽取的樣本
| Flower | Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|---|
| 1 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 2 | 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 3 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4 | 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5 | 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| 6 | 5.4 | 3.9 | 1.7 | 0.4 | setosa |
| 7 | 4.6 | 3.4 | 1.4 | 0.3 | setosa |
| 8 | 5.0 | 3.4 | 1.5 | 0.2 | setosa |
| 9 | 4.4 | 2.9 | 1.4 | 0.2 | setosa |
| 10 | 4.9 | 3.1 | 1.5 | 0.1 | setosa |
| 11 | 5.4 | 3.7 | 1.5 | 0.2 | setosa |
| 12 | 4.8 | 3.4 | 1.6 | 0.2 | setosa |
| 13 | 4.8 | 3.0 | 1.4 | 0.1 | setosa |
| 14 | 4.3 | 3.0 | 1.1 | 0.1 | setosa |
| 15 | 5.8 | 4.0 | 1.2 | 0.2 | setosa |
| 16 | 5.7 | 4.4 | 1.5 | 0.4 | setosa |
| 17 | 5.4 | 3.9 | 1.3 | 0.4 | setosa |
| 18 | 5.1 | 3.5 | 1.4 | 0.3 | setosa |
| 19 | 5.7 | 3.8 | 1.7 | 0.3 | setosa |
| 20 | 5.1 | 3.8 | 1.5 | 0.3 | setosa |
| 21 | 5.4 | 3.4 | 1.7 | 0.2 | setosa |
| 22 | 5.1 | 3.7 | 1.5 | 0.4 | setosa |
| 23 | 4.6 | 3.6 | 1.0 | 0.2 | setosa |
| 24 | 5.1 | 3.3 | 1.7 | 0.5 | setosa |
| 25 | 4.8 | 3.4 | 1.9 | 0.2 | setosa |
| 51 | 7.0 | 3.2 | 4.7 | 1.4 | versicolor |
| 52 | 6.4 | 3.2 | 4.5 | 1.5 | versicolor |
| 53 | 6.9 | 3.1 | 4.9 | 1.5 | versicolor |
| 54 | 5.5 | 2.3 | 4.0 | 1.3 | versicolor |
| 55 | 6.5 | 2.8 | 4.6 | 1.5 | versicolor |
| 56 | 5.7 | 2.8 | 4.5 | 1.3 | versicolor |
| 57 | 6.3 | 3.3 | 4.7 | 1.6 | versicolor |
| 58 | 4.9 | 2.4 | 3.3 | 1.0 | versicolor |
| 59 | 6.6 | 2.9 | 4.6 | 1.3 | versicolor |
| 60 | 5.2 | 2.7 | 3.9 | 1.4 | versicolor |
| 61 | 5.0 | 2.0 | 3.5 | 1.0 | versicolor |
| 62 | 5.9 | 3.0 | 4.2 | 1.5 | versicolor |
| 63 | 6.0 | 2.2 | 4.0 | 1.0 | versicolor |
| 64 | 6.1 | 2.9 | 4.7 | 1.4 | versicolor |
| 65 | 5.6 | 2.9 | 3.6 | 1.3 | versicolor |
| 66 | 6.7 | 3.1 | 4.4 | 1.4 | versicolor |
| 67 | 5.6 | 3.0 | 4.5 | 1.5 | versicolor |
| 68 | 5.8 | 2.7 | 4.1 | 1.0 | versicolor |
| 69 | 6.2 | 2.2 | 4.5 | 1.5 | versicolor |
| 70 | 5.6 | 2.5 | 3.9 | 1.1 | versicolor |
| 71 | 5.9 | 3.2 | 4.8 | 1.8 | versicolor |
| 72 | 6.1 | 2.8 | 4.0 | 1.3 | versicolor |
| 73 | 6.3 | 2.5 | 4.9 | 1.5 | versicolor |
| 74 | 6.1 | 2.8 | 4.7 | 1.2 | versicolor |
| 75 | 6.4 | 2.9 | 4.3 | 1.3 | versicolor |
| 101 | 6.3 | 3.3 | 6.0 | 2.5 | virginica |
| 102 | 5.8 | 2.7 | 5.1 | 1.9 | virginica |
| 103 | 7.1 | 3.0 | 5.9 | 2.1 | virginica |
| 104 | 6.3 | 2.9 | 5.6 | 1.8 | virginica |
| 105 | 6.5 | 3.0 | 5.8 | 2.2 | virginica |
| 106 | 7.6 | 3.0 | 6.6 | 2.1 | virginica |
| 107 | 4.9 | 2.5 | 4.5 | 1.7 | virginica |
| 108 | 7.3 | 2.9 | 6.3 | 1.8 | virginica |
| 109 | 6.7 | 2.5 | 5.8 | 1.8 | virginica |
| 110 | 7.2 | 3.6 | 6.1 | 2.5 | virginica |
| 111 | 6.5 | 3.2 | 5.1 | 2.0 | virginica |
| 112 | 6.4 | 2.7 | 5.3 | 1.9 | virginica |
| 113 | 6.8 | 3.0 | 5.5 | 2.1 | virginica |
| 114 | 5.7 | 2.5 | 5.0 | 2.0 | virginica |
| 115 | 5.8 | 2.8 | 5.1 | 2.4 | virginica |
| 116 | 6.4 | 3.2 | 5.3 | 2.3 | virginica |
| 117 | 6.5 | 3.0 | 5.5 | 1.8 | virginica |
| 118 | 7.7 | 3.8 | 6.7 | 2.2 | virginica |
| 119 | 7.7 | 2.6 | 6.9 | 2.3 | virginica |
| 120 | 6.0 | 2.2 | 5.0 | 1.5 | virginica |
| 121 | 6.9 | 3.2 | 5.7 | 2.3 | virginica |
| 122 | 5.6 | 2.8 | 4.9 | 2.0 | virginica |
| 123 | 7.7 | 2.8 | 6.7 | 2.0 | virginica |
| 124 | 6.3 | 2.7 | 4.9 | 1.8 | virginica |
| 125 | 6.7 | 3.3 | 5.7 | 2.1 | virginica |
該過程按以下步驟進行
#import data data <- read.table(“sampleiris.txt”) #execution result <- pam(data[1:4], 3, FALSE, “euclidean”) #print results summary(result) #plot clusters plot (data, col = result$clustering) #add the medoids to the plot points(result$medoids, col = 1:3, pch = 4)
執行結果中列印了以下資料。
Medoids:
ID Sepal.Length Sepal.Width Petal.Length Petal.Width
8 8 5.0 3.4 1.5 0.2
64 39 6.1 2.9 4.7 1.4
103 53 7.1 3.0 5.9 2.1
Clustering vector:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22
23 24 25 51 52 53 54 55 56
1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
1 1 1 2 2 2 2 2 2
57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 101 102 103
104 105 106 107 108 109 110 111 112
2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 3 2 3
3 3 3 2 3 3 3 2 2
113 114 115 116 117 118 119 120 121 122 123 124 125
3 2 2 3 3 3 3 2 3 2 3 2 3
Objective function:
build swap
0.7148339 0.6990539
Numerical information per cluster:
size max_diss av_diss diameter separation
[1,] 25 1.236932 0.5137400 2.042058 1.9000000
[2,] 34 1.951922 0.8085343 2.727636 0.3741657
[3,] 16 1.284523 0.7559609 2.147091 0.3741657
Isolated clusters:
L-clusters: [1] 1
L*-clusters: character(0)
Silhouette plot information:
cluster neighbor sil_width
1 1 2 0.84941732
5 1 2 0.84830238
8 1 2 0.84812593
18 1 2 0.84784555
12 1 2 0.83221128
22 1 2 0.82890349
20 1 2 0.82456328
3 1 2 0.82337894
7 1 2 0.81910409
10 1 2 0.81662688
11 1 2 0.80769429
2 1 2 0.80592613
13 1 2 0.80278163
4 1 2 0.79810574
23 1 2 0.79482977
24 1 2 0.78999596
17 1 2 0.78539723
21 1 2 0.78454015
25 1 2 0.77452963
6 1 2 0.75995941
9 1 2 0.74605493
14 1 2 0.74277337
19 1 2 0.72082914
15 1 2 0.71581750
16 1 2 0.66155611
68 2 3 0.60036142
56 2 3 0.59753885
62 2 3 0.59698924
72 2 3 0.59691421
70 2 3 0.59514179
54 2 3 0.58507022
67 2 3 0.56989428
60 2 1 0.56350914
63 2 3 0.55592514
75 2 3 0.54720666
74 2 3 0.53971473
64 2 3 0.53757677
69 2 3 0.51098390
65 2 1 0.50762488
107 2 3 0.48295375
55 2 3 0.46851074
52 2 3 0.46827948
59 2 3 0.44164146
66 2 3 0.42147865
71 2 3 0.41421605
73 2 3 0.41282512
122 2 3 0.40891392
120 2 3 0.40207904
57 2 3 0.39510378
114 2 3 0.37176468
124 2 3 0.34854822
102 2 3 0.33532624
61 2 1 0.32662688
58 2 1 0.20142024
51 2 3 0.19024422
115 2 3 0.16320750
53 2 3 0.11554863
112 2 3 -0.07433144
111 2 3 -0.07748205
103 3 2 0.59622203
106 3 2 0.59241159
108 3 2 0.58027197
110 3 2 0.56716967
123 3 2 0.56182697
121 3 2 0.55568135
119 3 2 0.53242285
118 3 2 0.52551154
125 3 2 0.51206488
105 3 2 0.49243542
101 3 2 0.45749953
113 3 2 0.44409513
109 3 2 0.37181492
117 3 2 0.26375026
116 3 2 0.21777715
104 3 2 0.21412781
Average silhouette width per cluster:
[1] 0.7931708 0.4153331 0.4678177
Average silhouette width of total data set:
[1] 0.5524757
2775 dissimilarities, summarized :
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.1000 1.1136 2.5080 2.6329 3.9006 7.0852
Metric : euclidean
Number of objects : 75
Available components:
[1] "medoids" "id.med" "clustering" "objective" "isolation" "clusinfo"
"silinfo" "diss" "call"
[10] "data"
同時生成了以下圖形。
在執行演算法並分析資料後,我們可以得出結論:聚類分組良好,與每朵花的種類相關。資料中共有 75 個元素,25 個屬於Setosa 種類,25 個屬於Versicolor 種類,25 個屬於Virginica 種類,演算法將Setosa 的元素聚類為叢集 1,將Versicolor 的元素聚類為叢集 2,將Virginica 的元素聚類為叢集 3。在驗證結果後,我們發現 75 個元素中,有 66 個被正確地聚類,誤差率為 12%——這是一個非常好的結果。
- R 開發核心團隊,R:一種統計計算語言和環境。
- Kaufman, L., Rousseeuw, P. J., 中位數聚類。