生命科學方法與概念/DNA測序
Allan Maxam和Walter Gilbert於1977年發表了一種基於DNA化學修飾和隨後在特定鹼基處切割的DNA測序方法。該方法也被稱為化學測序,允許使用純化的雙鏈DNA樣品,而無需進一步克隆。由於該方法使用放射性標記和其技術複雜性,在Sanger方法改進後,其使用率逐漸減少。
Maxam-Gilbert測序需要在DNA的5'末端進行放射性標記,並純化要測序的DNA片段。化學處理隨後在四種反應(G、A+G、C、C+T)中的一個或兩個中,在少量四種核苷酸鹼基中的一個或兩個處產生斷裂。控制修飾化學物質的濃度,以平均在每個DNA分子中引入一個修飾。因此,會生成一系列標記的片段,從放射性標記的末端到每個分子中的第一個“切割”位點。四種反應中的片段在變性丙烯醯胺凝膠中並排電泳,進行大小分離。為了視覺化這些片段,將凝膠暴露在X射線片中進行放射自顯影,從而產生一系列暗帶,每個暗帶對應一個放射性標記的DNA片段,由此可以推斷出序列。


Frederick Sanger和同事在1977年開發的鏈終止方法很快就成為首選方法,因為它相對容易和可靠。發明時,鏈終止方法比Maxam和Gilbert方法使用了更少的毒性化學物質和更少的放射性。由於其相對的易用性,Sanger方法很快實現了自動化,並且是第一代DNA測序儀中使用的方法。
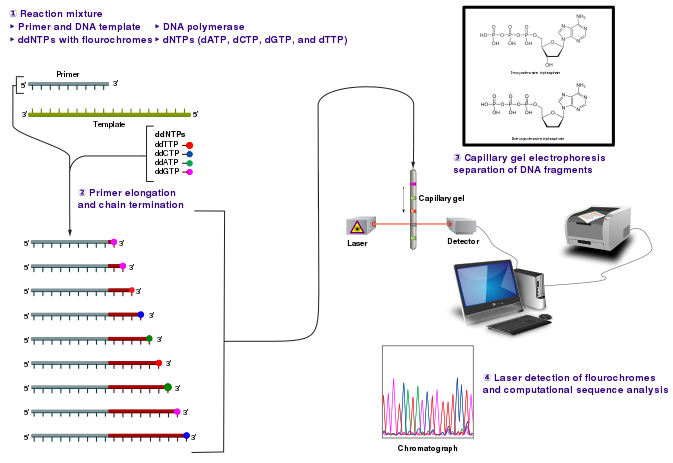
經典的鏈終止方法需要單鏈DNA模板、DNA引物、DNA聚合酶、正常脫氧核苷三磷酸 (dNTPs) 和修飾的雙脫氧核苷三磷酸 (ddNTPs),後者會終止DNA鏈延伸。這些鏈終止核苷酸缺乏形成兩個核苷酸之間磷酸二酯鍵所需的 3'-OH 基團,導致DNA聚合酶在摻入 ddNTP 後停止DNA延伸。
DNA樣品被分成四個獨立的測序反應,每個反應包含所有四種標準dNTPs和DNA聚合酶。在每個反應中只新增四種雙脫氧核苷酸 (ddATP、ddGTP、ddCTP或ddTTP) 中的一種。在模板DNA從結合的引物進行多輪延伸後,將所得的DNA片段進行熱變性,並使用凝膠電泳按大小分離。在1977年的最初出版物中,Sanger使用放射性標記的dATP來檢測條帶,方法是放射自顯影。
右側的影像顯示了一個暴露在測序凝膠中的X射線片,暗帶對應不同長度的DNA片段。一條泳道中的暗帶表示在摻入特定ddNTP後鏈終止產生的DNA片段。然後,使用四個泳道中不同條帶的相對位置(從下到上)來讀取DNA序列。
鏈終止測序的技術變體包括使用標記的引物,該引物可以是放射性的,也可以包含熒光染料。染料引物測序便於在光學系統中讀取,以實現更快、更經濟的分析和自動化。
迴圈測序將Sanger測序和PCR結合起來,對極少量的DNA進行測序。測序反應在熱迴圈儀中進行,其中耐熱聚合酶的重複變性、退火和DNA合成導致鏈終止產物的擴增。與正常的PCR相比,這種擴增不是指數型的,而是線性的,因為只使用一個引物。

染料終止子測序利用鏈終止ddNTPs的標記,允許在單一反應中進行測序,而不是像標記引物方法那樣進行四種反應。在染料終止子測序中,四種ddNTP鏈終止子中的每一種都用熒光染料標記,每種染料在不同的波長下發出光。
由於其更高的效率和速度,染料終止子測序現在是自動化測序中的主要方法。它的侷限性包括染料效應,這是由於染料標記的鏈終止子在DNA片段中摻入的差異導致的,從而導致毛細管電泳後電子DNA序列軌跡圖譜中峰高和形狀不一致。
這個問題已經透過使用修飾的DNA聚合酶酶系統和最大限度地減少摻入差異的染料,以及消除“染料斑點”的方法得到了解決。染料終止子測序方法以及自動化的高通量DNA測序儀現在被用於絕大多數測序專案。
使用Sanger方法進行DNA測序的常見挑戰包括序列前15-40個鹼基的質量較差(由於引物結合導致),以及在700-900個鹼基後測序軌跡質量下降。當前方法只能在單一反應中直接測序相對較短的DNA片段(長度為300-1000個核苷酸)。測序大於此尺寸限制的DNA片段的主要障礙是,對於長度僅相差一個核苷酸的大型DNA片段,分離能力不足。

焦磷酸測序方法於 1996 年開發,基於檢測使用化學發光酶的 DNA 聚合酶活性。本質上,該方法允許透過沿其合成互補鏈,一次一個鹼基對,並檢測在每個步驟中實際添加了哪個鹼基來對單鏈 DNA 進行測序。模板 DNA 是固定的,A、C、G 和 T 核苷酸的溶液依次新增和從反應中移除。只有當核苷酸溶液與模板的第一個未配對鹼基互補時才會產生光。產生化學發光訊號的溶液順序允許確定模板的順序。
將 ssDNA 模板與測序引物雜交,並與酶 DNA 聚合酶、ATP 硫酸化酶、熒光素酶和焦磷酸酶以及底物腺苷 5´ 磷酸硫酸鹽 (APS) 和熒光素一起溫育。
- 新增四種脫氧核苷三磷酸 (dNTP) 之一將啟動第二步。DNA 聚合酶將正確的互補 dNTP 摻入模板中。這種摻入會按化學計量釋放焦磷酸 (PPi)。
- 在存在腺苷 5´ 磷酸硫酸鹽的情況下,ATP 硫酸化酶定量地將 PPi 轉換為 ATP。這種 ATP 作為熒光素酶介導的熒光素轉換為氧化的熒光素的底物,在與 ATP 量成正比的量中產生可見光。在熒光素酶催化反應中產生的光被相機檢測並分析成焦磷酸圖譜。
- 未摻入的核苷酸和 ATP 被焦磷酸酶降解,反應可以使用另一種核苷酸重新開始。
目前,該方法的一個侷限性是,DNA 序列的單個讀數的長度在 300-500 個核苷酸附近,短於鏈終止方法(例如桑格測序)可獲得的 800-1000 個核苷酸。這會使基因組組裝過程變得更加困難,特別是對於包含大量重複 DNA 的序列而言。截至 2007 年,焦磷酸測序最常用於對已知近親序列的基因組進行重測序或測序。
焦磷酸測序的模板可以透過固相模板製備(鏈黴親和素包被的磁珠)和酶促模板製備(焦磷酸酶+核酸外切酶)來製備。因此,焦磷酸測序分為兩種型別;即固相焦磷酸測序和液相焦磷酸測序。
對低成本測序的迫切需求推動了高通量測序(或下一代測序)技術的發展,這些技術並行化了測序過程,同時產生了數千或數百萬個序列。高通量測序技術旨在降低 DNA 測序成本,超出標準染料終止方法所能達到的水平。在超高通量測序中,可能同時執行多達 500,000 個合成測序操作。
454 測序使用大規模平行焦磷酸測序系統,能夠在每次 10 小時執行中測序大約 400-600 兆鹼基的 DNA。
基因組 DNA 被分成更小的片段(300-800 個鹼基對)並拋光(在每個末端使其變鈍)。然後將短接頭連線到片段的末端。這些接頭提供了用於樣品庫片段的擴增和測序的引物序列。一個接頭(接頭 B)包含一個 5´-生物素標籤,用於將 DNA 庫固定到鏈黴親和素包被的珠子上。在切口修復後,釋放非生物素化鏈,並用作單鏈模板 DNA (sstDNA) 庫。評估 sstDNA 庫的質量,並透過滴定確定 PCR 所需的最佳量(每個珠子的 DNA 複製數)。
sstDNA 庫固定在珠子上。包含庫片段的珠子攜帶一個單一的 sstDNA 分子。將珠子結合的庫與擴增試劑在油包水混合物中乳化。每個珠子在其自己的微反應器中被捕獲,在那裡發生 PCR 擴增。這導致珠子固定、克隆擴增的 DNA 片段。
將單鏈模板 DNA 庫珠子新增到 DNA 珠子溫育混合物(包含 DNA 聚合酶)中,並將酶珠(包含硫酸化酶和熒光素酶)分層到 PicoTiterPlate 裝置上。將裝置離心以將珠子沉積到孔中。酶珠層確保 DNA 珠子在測序反應過程中保持定位在孔中。珠子沉積過程旨在最大限度地提高含有單個擴增庫珠子的孔的數量。
將裝載的 PicoTiterPlate 放入 Genome Sequencer FLX 儀器中。流體系統在板的孔上輸送測序試劑(包含緩衝液和核苷酸)。在測序執行期間,四種 DNA 核苷酸按固定順序依次新增到 PicoTiterPlate 上。在核苷酸流動期間,與每個珠子結合的 DNA 的數百萬個複製並行測序。當與模板鏈互補的核苷酸新增到孔中時,聚合酶會透過新增核苷酸來延伸現有的 DNA 鏈。新增一個(或多個)核苷酸會產生光訊號,該訊號被儀器中的 CCD 相機記錄。訊號強度與核苷酸的數量成正比;例如,在單個核苷酸流中摻入的同聚物延伸產生的訊號比單個核苷酸產生的訊號更大。但是,同聚物延伸的訊號強度僅在八個連續核苷酸之前呈線性,之後訊號迅速下降。資料以標準流程圖格式 (SFF) 檔案儲存,供下游分析使用。
2007 年 3 月下旬,羅氏診斷宣佈達成協議,以 1.549 億美元收購 454 Life Sciences。2013 年 10 月,羅氏宣佈將關閉 454,並在 2016 年年中停止支援該平臺。

Solexa(現為 Illumina 的一部分)成立於 1998 年,開發了一種基於可逆終止子的合成測序技術。在這種方法中,首先將 DNA 剪下,並將兩個接頭連線到片段的末端。然後將單鏈、接頭連線的片段隨機結合到流動槽的表面,並透過橋擴增進行擴增:流動槽被塗有與接頭相對應的寡核苷酸。因此,片段的遊離末端可以“橋接”到表面上的互補寡核苷酸,然後作為 DNA 聚合酶的引物。新合成的雙鏈橋的變性會留下兩個與表面連線的單鏈片段。重複延伸和變性會在整個流動槽中產生數百萬個獨特的簇(或 polonies,即聚合酶生成的菌落)。在下一步中,可以使用可逆終止子對這些菌落進行測序。這些是其 3'‑OH 基團被化學封鎖的核苷酸。此外,四種類型的核苷酸帶有不同的熒游標記。在測序的第一輪迴圈中,將所有四種核苷酸、引物和 DNA 聚合酶新增到流動槽中。在每個模板中,可以摻入單個核苷酸,其餘的被沖洗掉。現在,使用雷射激發熒游標記,並拍攝整個流動槽的高解析度影像。任何高於背景的訊號都標識了簇的物理位置,熒光發射標識了摻入了哪四個鹼基。接下來,去除染料和末端 3´ 阻斷劑,新增四種可逆終止子和聚合酶,開始下一輪迴圈。透過這種方式,可以一次確定一個鹼基的片段序列。最終,可以對數百萬個讀數進行比對,以得出原始 DNA 的序列。
Illumina 目前是下一代測序機領域的市場領導者。它提供了幾種版本的測序儀,其中 HiSeq 是功能最強大,最常用於對大型基因組進行測序的儀器。據 Illumina 稱,HiSeq X Ten 是第一個打破人類基因組 1000 美元門檻的測序平臺。它可以為每個流動槽產生高達 30 億個讀數,每個模板兩端的最大讀數長度為 150 bp。執行時間最長可達三天。MiSeq 是一種更快、更便宜的替代方案,每個流動槽有 2500 萬個讀數,每個模板兩端的最大讀數長度為 300 bp。
Ion Torrent Systems Inc.(現為 Life Technologies 所有)開發了一種基於使用標準測序化學,但具有新穎的半導體檢測系統的系統。這種測序方法基於檢測 DNA 聚合反應過程中釋放的氫離子,而不是其他測序系統中使用的光學方法。包含要測序的模板 DNA 鏈的微孔被單一型別的核苷酸淹沒。如果引入的核苷酸與引導模板核苷酸互補,它就會被摻入到正在生長的互補鏈中。這會導致氫離子的釋放,從而觸發超靈敏的離子感測器,表明發生了反應。如果模板序列中存在同聚物重複序列,則在單個迴圈中會摻入多個核苷酸。這會導致相應數量的氫釋放和成比例更高的電子訊號。

SMRT 測序基於合成測序方法。DNA 在零模波導 (ZMW) 中合成 - 這些是底部帶有捕獲工具的小型井狀容器。測序使用未修飾的聚合酶 (連線到 ZMW 底部) 和自由流動的熒游標記核苷酸進行,這些核苷酸存在於溶液中。這些井的結構使得只有在井底部發生的熒光會被檢測到。熒游標記在核苷酸整合到 DNA 鏈中後會從核苷酸上脫落,留下未修飾的 DNA 鏈。根據 SMRT 技術開發商太平洋生物科學公司,這種方法允許檢測核苷酸修飾(如胞嘧啶甲基化)。這是透過觀察聚合酶動力學實現的。這種方法可以產生 20,000 個核苷酸或更長的讀長,平均讀長為 5 千鹼基。
該方法基於讀取核苷酸透過與環糊精共價結合的 α-溶血素孔時的電訊號。透過奈米孔的 DNA 會改變其離子電流。這種變化取決於 DNA 序列的形狀、大小和長度。每種型別的核苷酸會阻斷透過孔的離子流不同的時間。該方法具有發展潛力,因為它不需要修飾的核苷酸,但是單核苷酸解析度尚未實現。
奈米孔測序發展的兩個主要領域是固態奈米孔測序和蛋白質奈米孔測序。蛋白質奈米孔測序利用膜蛋白複合物,例如 ∝‑溶血素和 MspA(分枝桿菌 smegmatis 孔蛋白 A),由於它們能夠區分單個核苷酸和核苷酸組,因此顯示出巨大前景。固態奈米孔測序利用合成材料,例如氮化矽和氧化鋁,由於其優異的機械效能以及熱穩定性和化學穩定性而被優先選擇。對於這種型別的測序,製造方法至關重要,因為奈米孔陣列可以包含數百個直徑小於 8 奈米的孔。
這個概念源於這樣一種想法:單鏈 DNA 或 RNA 分子可以透過電泳以嚴格的線性順序透過生物孔,該孔可以小於 8 奈米,並且可以檢測到,因為分子在透過孔時會釋放離子電流。該孔包含一個檢測區域,能夠識別不同的鹼基,每個鹼基在穿過孔時會產生不同的特定時間訊號,這些訊號對應於鹼基的順序,然後進行評估。在實施此過程時,需要注意的是,精確控制 DNA 透過孔的傳輸對於成功至關重要。各種酶,如核酸外切酶和聚合酶,已被用於透過將它們放置在孔的入口附近來調節此過程。
引物步進是一種用於測序 1.3 到 7 千鹼基之間 DNA 片段的方法。這些片段太長,無法使用鏈終止方法在單次序列讀取中測序。該方法透過將長序列分成幾個連續的短序列來實現。目標 DNA 可以是質粒插入片段、PCR 產物或代表基因組測序時間隙的片段。
該片段首先像較短片段一樣進行測序 - 使用通用引物或客戶指定的引物從每個末端進行測序。這應該會識別出前 1000 個(大約)鹼基。為了完全測序感興趣的區域,需要設計和合成新的引物 - 互補於已知序列的最後 20 個鹼基 - 以獲得連續的序列資訊。
這樣,測序的長的 DNA 的短部分就會沿著序列“步進”。該方法可用於測序整個染色體(因此稱為染色體步進)。
在鳥槍法測序中,DNA 被隨機分解成許多小片段,這些片段被測序以獲得讀長。透過執行多輪這種片段化和測序,獲得目標 DNA 的多個重疊讀長。然後,計算機程式利用不同讀長的重疊末端將它們組裝成一個連續的序列。
傳統的鳥槍法測序基於桑格測序法:這是大約 1995 年至 2005 年期間用於基因組測序的最先進技術。鳥槍法策略仍然應用於今天,但使用的是下一代測序。這些技術產生較短的讀長(從 25 到 500bp 不等),但在相對較短的時間內(大約一天)會產生數十萬甚至數百萬個讀長。這會導致高覆蓋率,但組裝過程在計算上要昂貴得多。由於資料量大,以及測序整個基因組所需的時間相對較短,這些技術遠遠優於桑格測序。
- Mardis, E.R., 2008. Next-generation DNA sequencing methods. Annu. Rev. Genomics Hum. Genet. 9, 387–402.
- Metzker, M.L., 2010. Sequencing technologies - the next generation. Nat. Rev. Genet. 11, 31–46.
- Sanger, F., Coulson, A.R., 1975. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase. J. Mol. Biol. 94, 441–8.
- Sanger, F., Nicklen, S., Coulson, A.R., 1977. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463‑7.
