生命科學方法與概念/重組
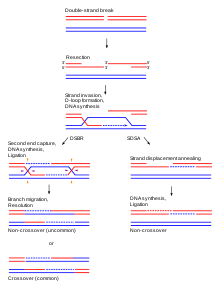
同源重組是一種遺傳重組,其中兩個相似或相同的 DNA 分子之間的核苷酸序列發生交換。它最常被細胞用來準確修復發生在 DNA 兩條鏈上的有害斷裂,稱為雙鏈斷裂。同源重組也產生減數分裂過程中 DNA 序列的新組合。此外,同源重組用於水平基因轉移,以交換細菌和病毒的不同菌株和物種之間的遺傳物質。
雖然同源重組在不同的生物體和細胞型別中差異很大,但大多數形式都涉及相同的基本步驟。在發生雙鏈斷裂後,斷裂處 5' 端周圍的 DNA 部分在稱為切除的過程中被切除。在接下來的鏈入侵步驟中,斷裂 DNA 分子的懸垂 3' 端然後“入侵”一個未斷裂的相似或相同的 DNA 分子。鏈入侵後,後續事件可能會遵循兩個主要途徑中的任何一個;DSBR(雙鏈斷裂修復)途徑或 SDSA(合成依賴性鏈退火)途徑。在 DNA 修復過程中發生的同源重組往往導致非交叉產物,實際上恢復了受損的 DNA 分子,就像雙鏈斷裂之前一樣。
同源重組也用於基因靶向,這是一種將遺傳改變引入目標生物體的技術。
Red/ET 是一種基於同源重組的商業重組技術。它涉及一個 λ噬菌體衍生的蛋白質對,Redα/Redβ,和 50 bp 同源區域。同源區域可以自由選擇,這意味著目標分子上的任何位置都可以改變。帶有可誘導啟動子的質粒用於表達重組蛋白。由於複製起點對溫度敏感,因此該質粒可以在重組後方便地移除。
在位點特異性重組中,DNA 鏈交換髮生在僅具有有限程度序列同源性的片段之間。位點特異性重組酶 (SSR) 透過識別和結合短 DNA 序列來執行 DNA 片段的重排,在該序列處它們切割 DNA 骨架,交換所涉及的兩個 DNA 螺旋並將 DNA 鏈重新連線起來。雖然在某些位點特異性重組系統中,僅僅重組酶和重組位點就足以執行所有這些反應,但在其他系統中,還需要一些輔助蛋白和/或輔助位點。多種基因組修飾策略,其中包括重組酶介導的盒式交換 (RMCE),一種針對將轉錄單元引入預定基因組位點的先進方法,依賴於 SSR 的能力。
位點特異性重組系統非常特異、快速和高效,即使面對複雜的真核基因組也是如此。它們被用於多種細胞過程,包括細菌基因組複製、分化和致病性以及移動遺傳元件的移動。出於同樣的原因,它們為開發基因工程工具提供了潛在的基礎。
重組位點通常在 30 到 200 個核苷酸之間,由兩個具有部分反向重複對稱性的基序組成,重組酶結合到這些基序上,並側翼重組發生的中心交叉序列。重組發生在其中的位點對通常是相同的,但也有例外。
根據氨基酸序列同源性和機制相關性,大多數位點特異性重組酶被歸類為兩個家族之一:酪氨酸重組酶家族或絲氨酸重組酶家族。這些名稱源於它們用於攻擊 DNA 並在此過程中與 DNA 共價連線的保守親核氨基酸殘基。
Cre/lox 重組被稱為位點特異性重組酶技術,被廣泛用於在細胞 DNA 的特定位點執行刪除、插入、易位和反轉。它在真核和原核系統中都有實現。
該系統由一種單一酶 Cre 重組酶組成,它重組一對稱為 lox 序列的短目標序列。該系統可以在不插入任何額外的支援蛋白或序列的情況下實現。Cre 酶和最初的Lox 位點稱為 loxP 序列源自噬菌體 P1。
將 Lox 序列適當地放置可以啟用、抑制基因或將基因交換為其他基因。Cre 酶的活性可以控制,以便它在特定細胞型別中表達或由外部刺激觸發,例如化學訊號或熱休克。這些有針對性的 DNA 改變在細胞譜系追蹤中以及當突變體在全球表達時是致命的時非常有用。
Cre/lox 系統在作用和使用方面與 FLP-FRT 重組系統非常相似。
Cre 重組酶(由最初命名為“Causes recombination”的基因座編碼,在某些參考文獻中發現“Cyclization recombinase”)是一種源自 P1 噬菌體的酪氨酸重組酶。該酶使用類似於拓撲異構酶 I 的機制來執行位點特異性重組事件。它由 4 個亞基和兩個結構域組成。總蛋白包含 343 個氨基酸。Cre 在 P1 噬菌體的生活週期中發揮著重要作用,例如線性基因組的環化和 DNA 複製後形成的二聚體染色體的解析。
LoxP(交叉點 P1 的基因座)是噬菌體 P1 上的一個位點,由 34 個 bp 組成。該位點包括一個不對稱的 8 bp 序列,除了中間兩個鹼基外,其他都是可變的,位於兩組迴文 13 bp 序列之間。下面給出了確切的序列;'N' 表示可能變化的鹼基。

當具有loxP位點的細胞在其基因組中表達Cre時,在loxP位點之間會發生重組事件。Cre重組酶與lox位點的第一個和最後一個13個鹼基對區域結合,形成二聚體。然後,該二聚體與另一個lox位點的二聚體結合形成四聚體。Lox位點是有方向性的,並且由四聚體連線的兩個位點在方向上是平行的。Cre蛋白在兩個loxP位點切割雙鏈DNA。然後,透過DNA連線酶將鏈重新連線,這是一個快速而有效的過程。
Cre介導的loxP位點重組的產物取決於loxP位點的位置和相對方向。位於兩個loxP位點之間的DNA序列被稱為“floxed”。在這種情況下,Cre介導的重組產物取決於loxP位點的方向。位於同一方向的兩個loxP位點之間的DNA將被切除,形成一個環狀DNA環,而位於相反方向的兩個loxP位點之間的DNA將被反轉。兩種包含loxP位點的獨立DNA物種可以透過Cre介導的重組進行融合。例如,可以使用變異的lox位點71和66連線質粒。
FLP/FRT
[edit | edit source]Flp-FRT重組是一種位點定向的重組技術,越來越多地用於在體內受控條件下操縱生物體的DNA。它類似於Cre-lox重組,但涉及由釀酒酵母2µm質粒衍生的重組酶(Flp)在短的翻轉酶識別靶標(FRT)位點之間進行序列重組。
34bp的最小FRT位點序列具有以下序列
5'GAAGTTCCTATTCtctagaaaGtATAGGAACTTC3'
翻轉酶(Flp)與兩個13bp的5'-GAAGTTCCTATTC-3'臂結合,該臂位於8bp間隔區兩側,即反向方向的位點特異性重組(交叉區域)。FRT介導的切割發生在頂鏈的非對稱8bp核心區域(5'tctagaaa3')前方,以及底鏈的該序列後面。存在多個變異的FRT位點,但重組通常只能在兩個相同的FRT之間發生,而不能在非相同(“異源性”)FRT之間發生。
許多可用的構建體包括一個額外的臂序列(5'-GAAGTTCCTATTCC-3'),該序列距離上游元件一個鹼基對,並且方向相同
5'GAAGTTCCTATTCcGAAGTTCCTATTCtctagaaaGtATAGGAACTTC3'
該片段對於切除是可有可無的,但對於整合是必不可少的,包括重組酶介導的盒交換。
由於重組活性可以靶向選定的器官,或者可以使用低水平的重組活性一致地改變僅一小部分細胞的DNA,因此Flp-FRT可用於在多細胞生物中構建遺傳鑲嵌體。使用這項技術,可以在感興趣的特定靶器官中研究基因的丟失或改變,即使在實驗動物無法承受其他器官中失去該基因的情況下也是如此(空間控制)。還可以透過使用誘導型啟動子在發育後期觸發重組活性來研究改變基因的效果(時間控制) - 這可以防止改變影響器官的整體發育,並允許在相同環境中比較缺乏該基因的單個細胞和正常的相鄰細胞。
應用
[edit | edit source]基因敲除
[edit | edit source]
基因敲除(縮寫:KO)是一種遺傳技術,其中生物體的一個基因被使其失活(“敲除”生物體)。它們也被稱為敲除生物或簡稱為敲除,用於瞭解已測序但功能未知或不完全已知的基因。研究人員從敲除生物體和正常個體之間的差異中得出推論。
敲除是透過多種技術的組合來完成的,從試管中的質粒、細菌人工染色體或其他DNA構建體開始,然後進行細胞培養。單個細胞被基因轉染了DNA構建體。通常目標是創造一種具有改變基因的轉基因動物。如果是這樣,胚胎幹細胞會被基因改造並插入早期胚胎。然後,具有其種系細胞中遺傳變化的產生的動物通常可以將基因敲除傳遞給後代。
構建體被設計用於與靶基因重組,這是透過將基因本身的序列整合到構建體中來實現的。然後在基因中該序列區域發生重組,導致插入外來序列以破壞基因。由於其序列被中斷,大多數情況下,改變的基因將被翻譯成無功能的蛋白質,如果它被翻譯的話。
因為在大多數細胞和大多數構建體的情況下,所需型別的DNA重組是一個罕見事件,所以選擇用於插入的外來序列通常包括一個報告基因。這使得能夠輕鬆選擇敲除成功的細胞或個體。有時DNA構建體插入染色體而不發生與靶基因的所需同源重組。為了消除這些細胞,DNA構建體通常包含第二個DNA區域,該區域允許識別和丟棄這些細胞。下面的圖示說明了這樣的系統。

在二倍體生物中,大多數基因包含兩個等位基因,並且可能包含幾個相關基因,它們在相同的作用中協同工作,因此需要進行額外的轉化和選擇輪次,直到每個靶基因都被敲除。可能需要選擇性育種才能產生純合敲除動物。
條件性敲除
[edit | edit source]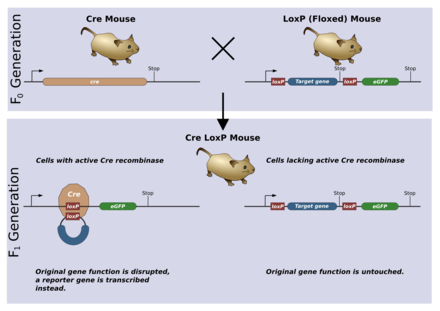
條件性敲除允許以組織或時間特異性方式刪除基因。這通常是透過使用Cre/lox系統來實現的。loxP序列將透過與正常敲除相同的機制被引入種系。然後,該種系可以與包含Cre重組酶基因的另一個種系雜交。通常,該基因將受組織特異性啟動子或僅在個體發育中特定時間活躍的啟動子控制。當Cre表達時,它會重組loxP位點並刪除floxed基因。
敲除主要用於透過比較敲除生物體與具有相似遺傳背景的野生型來了解特定基因或DNA區域的作用。
敲除生物體也被用作藥物開發中的篩選工具,透過使用特定的敲除來靶向特定的生物學過程或缺陷,或者透過使用涵蓋整個基因組的敲除生物體庫來了解藥物的作用機制,例如在釀酒酵母中。
敲入類似於敲除,但它用另一個基因代替了基因,而不是刪除它。
生產敲除小鼠的過程
[edit | edit source]生產敲除小鼠的過程有多種變異;以下是一個典型的例子。


- 要敲除的基因是從小鼠基因庫中分離出來的。然後,構建一個新的DNA序列,該序列與原始基因及其直接相鄰序列非常相似,只是它被改變得足以使基因失活。通常,新的序列還賦予了一個標記基因,該基因是正常小鼠沒有的,並賦予對某種有毒物質的抗性或產生可觀察到的變化(例如,顏色或熒光)。
- 從小鼠囊胚(非常年輕的胚胎)中分離出幹細胞,並在體外培養。對於本例,我們將取自白鼠的幹細胞。
- 透過電穿孔將步驟1中的新序列引入步驟2中的幹細胞。透過同源重組的自然過程,一些電穿孔的幹細胞會將其染色體中的原始基因替換為新的序列,其中包含敲除的基因。成功重組事件的機率相對較低,因此大多數改變的細胞在新序列中只有一個相關染色體 - 據說它們是雜合的。
- 使用步驟1中的標記基因將包含敲除基因的幹細胞從未改變的細胞中分離出來。例如,可以使用未改變的細胞對有毒物質敏感,而改變的細胞對該有毒物質有抗性。
- 將步驟4中的敲除幹細胞插入小鼠囊胚。對於本例,我們使用來自灰鼠的囊胚。囊胚現在包含兩種型別的幹細胞:原始幹細胞(來自灰鼠)和敲除幹細胞(來自白鼠)。然後將這些囊胚植入雌鼠的子宮中,在那裡它們發育。因此,新生的小鼠將是嵌合體:它們身體的某些部分來自原始幹細胞,其他部分來自敲除幹細胞。它們的毛皮將顯示出白色和灰色的斑塊,白色斑塊來自敲除幹細胞,灰色斑塊來自受體囊胚。
- 一些新生嵌合小鼠的性腺將來自敲除的幹細胞,因此會產生包含敲除基因的卵子或精子。當這些嵌合小鼠與野生型小鼠雜交時,它們的後代中會有一些在其所有細胞中都包含敲除基因的一個複製。這些小鼠將完全是白色的,並且不是嵌合體,但它們仍然是雜合子。
- 當這些雜合子後代相互交配時,它們的後代中會有一些會從父母雙方遺傳到敲除基因;它們沒有原始未改變基因的功能性複製(即,它們是該等位基因的純合子)。
