下一代測序 (NGS)/預處理
FASTQ 檔案透過包含序列質量資訊來擴充套件 FASTA 檔案中的序列資訊。FASTQ 檔案通常包含四行。
- 以 @ 開頭的行,包含序列識別符號
- 實際序列
- 以 + 開頭的行,後跟可選序列識別符號
- 用 ASCII 空間編碼的質量值行
因此,第二行和第四行必須具有相同的長度。下面給出一個示例,顯示一個序列“ATGTCT”...
@HWI-ST999:102:D1N6AACXX:1:1101:1235:1936 1:N:0: ATGTCTCCTGGACCCCTCTGTGCCCAAGCTCCTCATGCATCCTCCTCAGCAACTTGTCCTGTAGCTGAGGCTCACTGACTACCAGCTGCAG + 1:DAADDDF<B<AGF=FGIEHCCD9DG=1E9?D>CF@HHG??B<GEBGHCG;;CDB8==C@@>>GII@@5?A?@B>CEDCFCC:;?CCCAC
這裡將 -5 到 41 範圍內的質量值新增到偏移量,然後從 ASCII 表中獲取結果字元。因此,整個資料可以表示為文字。雖然 Illumina 對質量格式進行了多次更改,最終幾乎恢復到 Sanger 編碼,但最重要的是偏移量是 33(如 Sanger 和 Illumina v1.8 及更高版本)還是 64(如以前的 Illumina(和 Solexa)格式)。從圖表中可以看到,如果你發現以下任何字元:! "# $ % & ' ( ) * + , - . / 0 1 2 3 4 5 6 7 8 9 :,你的偏移量必須是 33,而以下任何字元 KLMNOPQRSTUVWXYZ[\]^_`abcdefgh 指向偏移量 64。上面的示例因此以 33 為基準偏移,因為我們發現第一個字元是 1。還要記住 @ 和 + 符號是質量的有效字元,因此即使一行以 @ 或 + 開頭,這也可能只是質量字串的開頭。
請參閱下面的質量圖表,該圖表是根據 維基百科 文章修改的。
SSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSS..................................................... ..........................XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX...................... ...............................IIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIII...................... .................................JJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJ...................... LLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLL.................................................... !"#$%&'()*+,-./0123456789:;<=>?@ABCDEFGHIJKLMNOPQRSTUVWXYZ[\]^_`abcdefghijklmnopqrstuvwxyz{|}~ | | | | | | 33 59 64 73 104 126 0........................26...31.......40 -5....0........9.............................40 0........9.............................40 3.....9.............................40 0........................26...31........41
S - Sanger and Illumina 1.8+, Offset 33, Quality range (0, 40) (!,I) X - Solexa, Offset 64, Quality range (-5, 40) (;,h) I - Illumina 1.3+ Offset 64, Quality range (0, 40) (@,h) J - Illumina 1.5+ Offset 64, Quality range (3, 40) (B,h) L - Illumina 1.8+ Offset 33, Quality range (0, 41) (!,J)
當進行質量控制時,需要考慮各種指標。
最簡單的方法是顯然是上面 FASTQ 檔案中介紹的質量評分。因此,它已經給出了關於鹼基呼叫質量的有效想法。由於讀段的質量通常在序列過程中逐漸下降,因此通常的做法是透過對檔案中所有讀段的質量進行平均來確定第一個、第二個、第三個、...第 n 個鹼基的平均質量。此外,為了瞭解一些關於分佈的資訊,通常會給出顯示分位數的條形圖。這將讓我們瞭解資料需要進行哪種修剪和質量過濾。
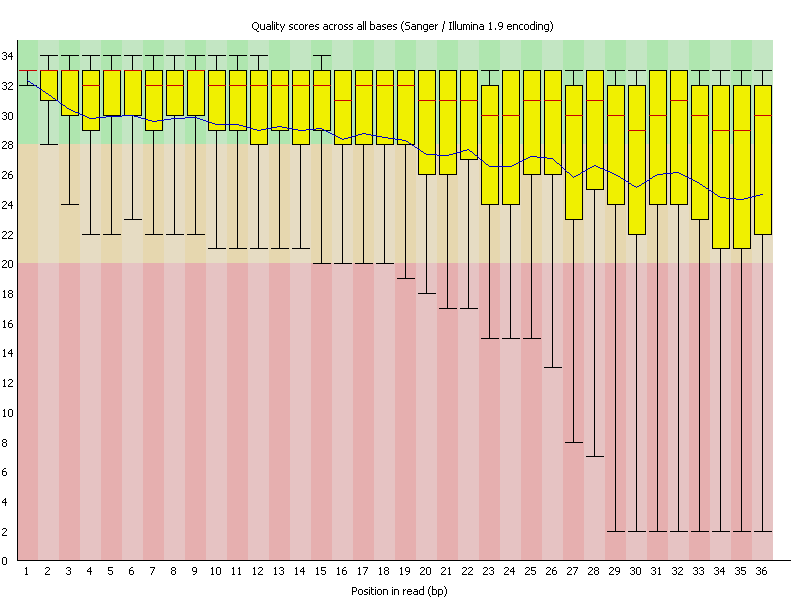
例如,這裡使用 FastQC 對序列資料進行了質量調查。可以看到,序列讀段長 36 個鹼基,平均序列質量(用藍色線表示)穩步下降。在許多新的 Illumina 試劑盒中,序列質量會在穩步下降之前先上升一點。
但是,除了檢查每個鹼基之外,還可以對每個讀段的質量進行平均,並顯示這些序列質量的累積圖。
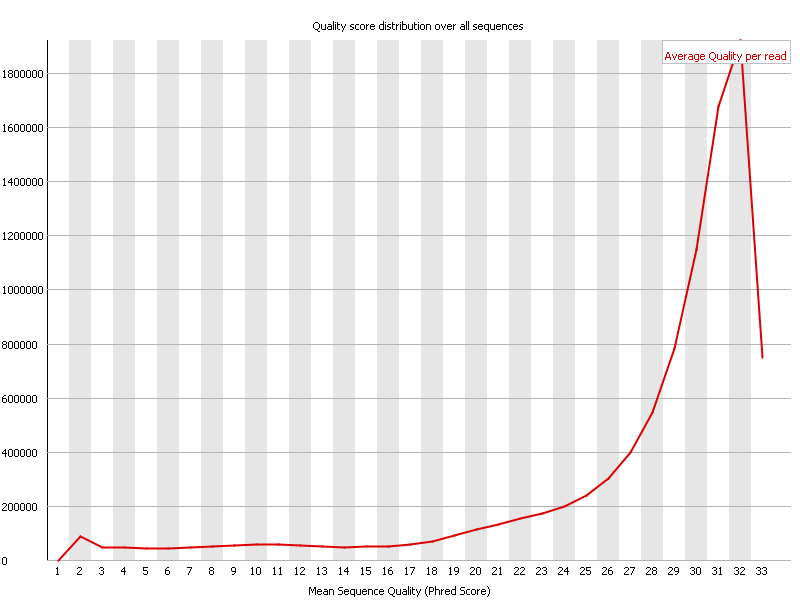
在上面的螢幕截圖中,可以看到大多數讀段的平均質量為 32。這通常被認為是非常好的,但是鑑於這些讀段有點短,它可能充其量只是中等水平的結果。
另一個重要的指標是檢視每個位置的鹼基內容。假設資料是從序列空間中隨機取樣的,則在每個位置,貢獻應該是相同的。因此,需要看到直線。實際上,第一個鹼基有時會表現出一些不規則的行為,這可能是由於引物不完全隨機造成的。但在所示示例中,讀段完全偏離了。可以看到,在整個讀段中,每個鹼基的偏差都很大。事實上,這種偏差非常強烈,你甚至可以讀出讀段中過度表示的鹼基。

例如,如果你檢視最後幾個鹼基,你可以將它們讀為 CTTGAAA - 序列結束。
如果我們現在將注意力轉向 FastQC 中過度表示的序列,我們可以立即找出原因
| 序列 | 計數 | 百分比 | 可能來源 |
|---|---|---|---|
| GATCGGAAGAGCTCGTATGCCGTCTTCTGCTTGAAA | 1870684 | 19.446980406066654 | Illumina 單端介面卡 1(33bp 上的 100%) |
| GAAGAGCTCGTATGCCGTCTTCTGCTTGAAAAAAAA | 95290 | 0.9906017065918623 | Illumina 單端介面卡 1(28bp 上的 100%) |
如上所述,鹼基呼叫器分配一個質量評分,該評分隨後可用於每個鹼基。這提供了對該鹼基可靠性的估計。請注意,取決於你的測序平臺,典型的錯誤型別不同。Illumina 最常見的錯誤型別是核苷酸交換,而 454、Ion Torrent/Proton 和其他類似平臺在同聚物(例如 AAAAAA)方面存在重大問題,其中正確數量的 A 往往無法完全確定。
為了處理較低的質量資料,通常會去除低質量鹼基。通常,會使用滑動視窗方法從例如 3' 端去除低質量鹼基,因為每個鹼基的質量逐漸下降。
除了去除低質量鹼基資料之外,還會去除介面卡、PCR 引物和其他偽影。在實踐中,會將介面卡剪下與質量修剪方法結合起來。
使用kmer方法糾正鹼基錯誤有不同的方法,因為有些錯誤無法簡單地剪下掉,即使質量值很高,也不能保證讀段沒有錯誤。這是在組裝之前的一個重要步驟,但對於比對來說可能不太重要。
一個基本的想法是基於讀段字串中的kmers。最初的想法可以追溯到至少2001年(Pevzner 2001),首先生成一個kmer譜,然後將超過一定閾值的kmers(稱為“solid”)和低於此閾值的kmers視為潛在的錯誤。
如果一個讀段被分成多個kmers,一個測序錯誤會導致幾個重疊的kmers從強變弱。錯誤校正步驟現在可以嘗試找到使讀段中所有kmers變強的最小更改次數。
像Quake這樣的變體也考慮了鹼基質量,以便更好地區分低複製真實kmers和高複製錯誤kmers。
數字歸一化和分割槽
[edit | edit source]特別是考慮到RNA測序,眾所周知,使用分子生物學技術(實驗室歸一化)對RNA進行歸一化可以幫助提供更好的拼接結果,並且可以獲得更好的總體代表性。這是因為使用實驗室歸一化會耗盡常見或高表達的序列。因此,在進行序列空間取樣時,在實驗室歸一化後,找到以前非常常見的序列的可能性較小,而找到以前表達不足的序列的可能性更大。除了找到表達不足的序列的可能性更高外,還有一個額外的好處,即由於過度取樣,現在不太可能在兩個或多個獨立讀段中多次找到相同的測序錯誤。後者使得組裝軟體不太可能錯誤地從一個真正的mRNA中建立多個拼接結果,這是由於這些相關的SNP造成的。也就是說,實驗室歸一化既不容易,如果外包出去,成本很高。因此,可以使用數字歸一化來替代。基本思想是下采樣具有大量豐富kmers的讀段。此外,這還有助於減少需要處理的讀段數量,因此組裝轉錄組可能更加可行(和更快)。一種實現方法是使用Titus Brown的工具集:http://ged.msu.edu/papers/2012-diginorm/
配對末端合併
[edit | edit source]許多工具將使用Illumina雙端測序資料,並在它們之間檢測到重疊時合併讀段,透過在不一致的位置採用更高質量的鹼基呼叫來糾正錯誤。這可以透過減少資料複雜性來提高組裝器的效能,並且還可以透過去除錯誤資料和改善重複序列的組裝來提高拼接結果。完成此操作的工具包括COPE 和FLASH.
去除其他不需要的序列
[edit | edit source]根據實驗的設計,在組裝之前可能還需要從讀段中去除或遮蔽其他不需要的序列。例如,如果對BAC或cosmid DNA池進行測序,可能需要去除大部分甚至所有載體骨架。類似地,大腸桿菌序列會汙染BAC或cosmid DNA製備物,可以在預先去除。也可以在組裝後去除這些序列。PhiX控制病毒DNA是Illumina測序資料中常見的汙染物。SMALT等快速搜尋工具可以用來將讀段對映到參考基因組,以便識別需要去除的讀段。
練習
[edit | edit source]QC工作流程(機器資料 -> 修剪前質量圖 -> 修剪 -> 修剪後質量圖)
[edit | edit source]因此,一個典型的工作流程可能是先從機器上獲取資料,然後評估上一節中所示的典型質量圖。這提供了對讀段質量的有效且重要的見解,並可能引起人們對可能發生的文庫製備問題的注意。在識別和記錄這些問題後,可以使用Trimmomatic等修剪工具去除一些錯誤,從序列末端去除低質量鹼基,(可能更重要的是)從讀段中去除剩餘的接頭等。在處理完讀段後,再次評估質量,以瞭解剩餘的質量問題。例如,即使在從序列中去除已知接頭(如上述情況)後,仍然可能觀察到每個鹼基的序列偏差,並且可能希望去除這種偏差或至少將其牢記在心。我們將在此討論一個示例工作流程。
資料集
[edit | edit source]從SRA下載SRR074262(這是上面的示例)。
Fastq輸出分析
[edit | edit source]下載FastQC(或在RobiNA中分析您的資料以獲得類似的圖)。FastQC比較容易理解。開啟剛剛下載的FastQ檔案。FastQC將執行您的資料集,您只需點選左側的各個類別,即可生成介紹中所示的圖。
僅去除接頭
[edit | edit source]我們將使用Trimmomatic簡單地去除接頭。java -jar trimmomatic-0.30.jar SE -phred33 SRR074262.fastq aclipped.fq.gz ILLUMINACLIP:TruSeq2-SE.fa:2:30:10 MINLEN:25 這告訴trimmomatic質量編碼是phred 33(現代Illumina),並將結果儲存在壓縮檔案adapter_clipped.fq.gz中。最後,它將使用trimmomatic提供的TruSeq3接頭。
Trimmomatic報告
輸入讀段:9619406 生存:7443332 (77.38%) 丟棄:2176074 (22.62%) TrimmomaticSE:成功完成
FastQC會自動識別gz檔案,因此壓縮檔案是可以的。實際上,當我們在FASTQC中開啟aclipped.fq.gz檔案時,接頭已被去除。
| 序列 | 計數 | 百分比 | 可能來源 |
|---|---|---|---|
| GGGGAGGAGAGAGCCATTGTTGAGGCGGCCATTGAG | 46910 | 0.630228505190954 | 無匹配 |
| AGGGGAGGAGAGAGCCATTGTTGAGGCGGCCATTGA | 21029 | 0.28252132244000405 | 無匹配 |
去除接頭和質量剪下
[edit | edit source]我們將使用Trimmomatic去除接頭,並使用滑動視窗方法去除末端的低質量鹼基。
java -jar trimmomatic-0.30.jar SE -phred33 SRR074262.fastq aclippedtrim.fq.gz ILLUMINACLIP:TruSeq2-SE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:25
輸入讀段:9619406 生存:6513310 (67.71%) 丟棄:3106096 (32.29%) TrimmomaticSE:成功完成
