生物化學原理/核酸 I:DNA 及其核苷酸
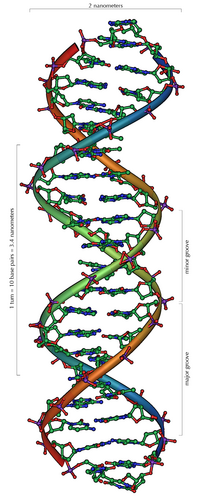
DNA 是一種由稱為核苷酸的重複單元組成的長聚合物。正如詹姆斯·D·沃森和弗朗西斯·克里克首次發現的那樣,所有物種的 DNA 結構都包含兩條螺旋鏈,每條螺旋鏈都圍繞同一個軸盤繞,每條螺旋鏈的螺距為 34 埃 (3.4 奈米),半徑為 10 埃 (1.0 奈米)。根據另一項研究,當在特定溶液中測量時,DNA 鏈的寬度為 22 到 26 埃 (2.2 到 2.6 奈米),一個核苷酸單元的長度為 3.3 埃 (0.33 奈米)。雖然每個重複單元都很小,但 DNA 聚合物可以是包含數百萬個核苷酸的超大分子。例如,人類最大的染色體,1 號染色體,大約有 2.2 億個鹼基對長。在生物體中,DNA 通常不以單分子的形式存在,而是以一對緊緊結合在一起的分子形式存在。這兩條長鏈像藤蔓一樣纏繞在一起,形成雙螺旋的形狀。核苷酸重複序列包含構成分子骨架的部分,該部分將鏈連線在一起,以及一個鹼基,該鹼基與螺旋中另一條 DNA 鏈相互作用。與糖連線的鹼基稱為核苷,而與糖和一個或多個磷酸基團連線的鹼基稱為核苷酸。如果多個核苷酸連線在一起,就像在 DNA 中一樣,這種聚合物稱為多核苷酸。DNA 鏈的骨架由交替的磷酸和糖殘基構成。DNA 中的糖是 2-脫氧核糖,這是一種戊糖(五碳)糖。糖透過形成相鄰糖環的第三和第五個碳原子之間的磷酸二酯鍵的磷酸基團連線在一起。這些不對稱鍵意味著 DNA 鏈有一個方向。在雙螺旋中,一條鏈中核苷酸的方向與另一條鏈中核苷酸的方向相反:鏈是反平行的。DNA 鏈的不對稱末端稱為 5'(5 質子)和 3'(3 質子)末端,其中 5' 末端具有末端磷酸基團,而 3' 末端具有末端羥基基團。DNA 和 RNA 之間的一個主要區別在於糖,DNA 中的 2-脫氧核糖被 RNA 中的另一種戊糖核糖取代。[1][2]
DNA 雙螺旋結構主要由兩種力量穩定:核苷酸之間的氫鍵和芳香族鹼基之間的鹼基堆積相互作用。在細胞的水環境中,核苷酸鹼基的共軛 π 鍵垂直於 DNA 分子的軸線排列,最大限度地減少了它們與溶劑化殼層的相互作用,從而最大限度地減少了吉布斯自由能。DNA 中發現的四種鹼基是腺嘌呤(縮寫為 A)、胞嘧啶(C)、鳥嘌呤(G)和胸腺嘧啶(T)。這四種鹼基與糖/磷酸基團連線形成完整的核苷酸,如腺嘌呤單磷酸所示。這些鹼基分為兩類;腺嘌呤和鳥嘌呤是融合的五元和六元雜環化合物,稱為嘌呤,而胞嘧啶和胸腺嘧啶是六元環,稱為嘧啶。第五種嘧啶鹼基,稱為尿嘧啶(U),通常在 RNA 中代替胸腺嘧啶,它與胸腺嘧啶的不同之處在於其環上缺少一個甲基。尿嘧啶通常不存在於 DNA 中,僅作為胞嘧啶的分解產物出現。除了 RNA 和 DNA 之外,還建立了許多人工核酸類似物,用於研究核酸的性質或用於生物技術。[3][4]


格里菲斯實驗由弗雷德里克·格里菲斯於 1928 年進行,是首批表明細菌能夠透過稱為轉化的過程轉移遺傳資訊。格里菲斯使用了兩種能夠感染小鼠的肺炎鏈球菌菌株——III-S 型(光滑型)和 II-R 型(粗糙型)菌株。III-S 型菌株用多糖莢膜包裹自身,該莢膜保護它免受宿主的免疫系統攻擊,導致宿主死亡,而 II-R 型菌株沒有這種保護性莢膜,被宿主的免疫系統擊敗。德國細菌學家弗雷德·諾伊費爾德發現了三種肺炎球菌型別(I 型、II 型和 III 型),並發現了 Quellung 反應,以便在體外識別它們。在格里菲斯實驗之前,細菌學家認為這些型別是固定的,從一代到下一代不可改變。在這個實驗中,III-S 型菌株中的細菌被熱殺死,它們的殘骸被新增到 II-R 型菌株細菌中。雖然單獨兩者都不會傷害小鼠,但它們的組合卻能夠殺死其宿主。格里菲斯還能夠從小鼠的血液中分離出活的 II-R 型和活的 III-S 型肺炎球菌。格里菲斯得出結論,II-R 型被來自已死 III-S 型菌株細菌的某種“轉化因子”轉化成了致命的 III-S 型菌株。今天,我們知道格里菲斯觀察到的“轉化因子”是 III-S 型菌株細菌的 DNA。雖然細菌被殺死了,但 DNA 卻在加熱過程中存活了下來,並被 II-R 型菌株細菌吸收。III-S 型菌株 DNA 包含形成保護性多糖莢膜的基因。由於擁有了這個基因,以前屬於 II-R 型的細菌現在可以免受宿主免疫系統的攻擊,並且可以殺死宿主。轉化因子的確切性質(DNA)在艾弗裡、麥克勞德和麥卡錫以及赫爾希和蔡斯進行的實驗中得到了驗證。[5]}}
阿爾弗雷德·赫爾希和瑪莎·蔡斯在 1952 年進行了一系列實驗,證實 DNA 是遺傳物質,這在 1944 年的艾弗裡-麥克勞德-麥卡錫實驗中首次得到證明。這些實驗被稱為赫爾希-蔡斯實驗。生物學家從 1869 年起就知道 DNA 的存在,但他們中的大多數人當時認為蛋白質承載著遺傳資訊。赫爾希和蔡斯在 T2噬菌體上進行了他們的實驗。噬菌體由一個包含其遺傳物質的蛋白質外殼組成。噬菌體透過附著在細菌的外膜上並注入其遺傳物質來感染細菌,留下其空殼附著在細菌上。
在他們的第一組實驗中,赫爾希和蔡斯用放射性磷-32 (p32) 對噬菌體的 DNA 進行了標記(磷元素存在於 DNA 中,但不存在於構成蛋白質的 20 種氨基酸中的任何一種)。他們讓噬菌體感染大腸桿菌,並透過一些巧妙的實驗,他們能夠觀察到標記有 P32 的噬菌體 DNA 轉移到細菌的細胞質中。在他們的第二組實驗中,他們用放射性硫-35 對噬菌體進行了標記(硫存在於氨基酸半胱氨酸和蛋氨酸中,但不存在於 DNA 中)。在感染大腸桿菌後,他們使用高速攪拌器將病毒蛋白外殼從感染的細胞上剪斷,並使用離心機分離細胞和病毒外殼。分離後,在蛋白質外殼中觀察到放射性 S35 示蹤劑,但在感染的細菌中沒有觀察到,這支援了以下假設:感染細菌的遺傳物質是 DNA 而不是蛋白質。[6][7] 赫爾希因其“關於病毒遺傳結構的發現”而分享了 1969 年的諾貝爾生理學或醫學獎。
奧斯瓦爾德·T·艾弗裡、科林·麥克萊德、麥克林·麥卡錫以及弗朗西斯·克里克和詹姆斯·D·沃森 [8]


兩條螺旋鍊形成DNA骨架。透過追蹤鏈之間的間隙或凹槽,可以找到另一條雙螺旋。這些空隙與鹼基對相鄰,可以提供一個結合位點。由於鏈不是直接相對的,因此凹槽的大小不一。一個凹槽,主溝,寬度為 22 Å,另一個,小溝,寬度為 12 Å。小溝的狹窄意味著鹼基的邊緣在主溝中更容易接近。因此,像轉錄因子這樣的蛋白質可以結合到雙鏈DNA中的特定序列中,通常與主溝中暴露的鹼基側面形成接觸。這種情況在細胞內DNA的異常構象中有所不同,但主溝和小溝的命名始終反映瞭如果DNA被扭回到普通的B形式,將會看到的尺寸差異。[9]。
查伽夫規則由埃爾溫·查伽夫提出,該規則指出,所有生物體任何細胞的DNA都應該具有嘧啶和嘌呤鹼基的 1:1 比例,更具體地說,鳥嘌呤的量等於胞嘧啶,腺嘌呤的量等於胸腺嘧啶。這種模式在DNA的兩條鏈中都可以找到。它們是由奧地利化學家埃爾溫·查伽夫發現的。
在分子生物學中,透過氫鍵連線在互補的DNA鏈上的兩個核苷酸被稱為鹼基對(通常縮寫為bp)。在規範的沃森-克里克DNA鹼基配對中,腺嘌呤 (A) 與胸腺嘧啶 (T) 形成鹼基對,鳥嘌呤 (G) 與胞嘧啶 (C) 形成鹼基對。在RNA中,胸腺嘧啶被尿嘧啶 (U) 取代。其他氫鍵模式,如擺動鹼基對和胡斯根鹼基對,也會出現——特別是在RNA中——從而產生複雜的功能性三級結構。[10]
例子
5'CTCGTTTGCGCTCTATCG3' 3'GAGCAAACGCGAGATAGC5'
1884年,德國化學家埃米爾·費歇爾給“嘌呤”命名(purum uricum)。他於1899年首次從尿酸中合成它,尿酸是由謝勒於1776年從腎結石中分離出來的。除了DNA和RNA之外,嘌呤也是許多其他重要生物分子中的成分,例如ATP、GTP、環AMP、NADH和輔酶A。嘌呤本身在自然界中沒有被發現,但它可以透過有機合成來製備。嘌呤是一種雜環芳香族有機化合物,由一個嘧啶環與一個咪唑環稠合而成。
例子
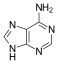
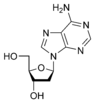
腺嘌呤是形成核酸(DNA或RNA)核苷酸的兩種嘌呤核鹼基之一(另一種是鳥嘌呤)。在DNA中,腺嘌呤透過兩個氫鍵與胸腺嘧啶結合,以幫助穩定核酸結構。腺嘌呤在連線到核糖時形成腺苷,一種核苷,在連線到脫氧核糖時形成脫氧腺苷。當三個磷酸基團新增到腺苷時,它形成腺苷三磷酸 (ATP),一種核苷酸。
鳥嘌呤與腺嘌呤和胞嘧啶一起存在於DNA和RNA中,而胸腺嘧啶通常只在DNA中看到,而尿嘧啶只在RNA中看到。在DNA中,鳥嘌呤與胞嘧啶配對。鳥嘌呤的化學式為C5H5N5O,是嘌呤的衍生物,由一個稠合的嘧啶-咪唑環體系組成,具有共軛雙鍵。
鳥嘌呤有兩種互變異構體形式,主要的酮式形式和稀有的烯醇式形式。它透過三個氫鍵與胞嘧啶結合。在胞嘧啶中,氨基作為氫供體,C-2羰基和N-3胺作為氫鍵受體。鳥嘌呤在C-6處有一個基團作為氫受體,而N-1處的基團和C-2處的氨基作為氫供體。



嘧啶是一種雜環芳香族有機化合物,類似於苯和吡啶,在六元環的第 1 位和第 3 位包含兩個氮原子。它與二嗪的另外兩種形式是異構體。核酸中發現的三個核鹼基,胞嘧啶 (C)、胸腺嘧啶 (T) 和尿嘧啶 (U),是嘧啶衍生物。
嘧啶與吡啶有很多共同的性質,隨著環中氮原子數量的增加,環π電子變得不那麼活躍和親電,親電芳香族取代變得更加困難,而親核芳香族取代變得更容易。最後一個反應型別的例子是2-氨基嘧啶中氨基被氯取代及其逆反應。嘧啶的共振穩定性降低可能導致加成和開環反應而不是取代反應。在迪姆羅斯重排中觀察到這種表現。與吡啶相比,N-烷基化和N-氧化更加困難,嘧啶的鹼性也更低:質子化的嘧啶的pKa值為1.23,而吡啶的pKa值為5.30。[11] 嘧啶也存在於隕石中,但科學家仍然不知道它的起源。嘧啶在紫外光下也能光解成尿嘧啶。

胞嘧啶可以作為DNA的一部分、RNA的一部分或核苷酸的一部分被發現。作為胞苷三磷酸 (CTP),它可以作為酶的輔因子,並且可以轉移一個磷酸基團將二磷酸腺苷 (ADP) 轉換為三磷酸腺苷 (ATP)。胞嘧啶的核苷是胞苷。在DNA和RNA中,胞嘧啶與鳥嘌呤配對。然而,它本質上是不穩定的,並且可以變成尿嘧啶(自發脫氨)。如果未被DNA修復酶(如尿嘧啶糖基化酶)修復,這會導致點突變,尿嘧啶糖基化酶可以切割DNA中的尿嘧啶。
胞嘧啶也可以被一種叫做DNA甲基轉移酶的酶甲基化為5-甲基胞嘧啶,或者被甲基化和羥基化以製備5-羥甲基胞嘧啶。胞嘧啶或5-甲基胞嘧啶的活性酶促脫氨作用,由APOBEC家族的胞嘧啶脫氨酶完成,可能對各種細胞過程以及有機體進化產生有益和有害的影響。另一方面,脫氨作用對5-羥甲基胞嘧啶的影響尚不清楚。[12]
胸腺嘧啶 (T, Thy) 是DNA核酸中四種核鹼基之一,用字母 G-C-A-T 表示。其他三種是腺嘌呤、鳥嘌呤和胞嘧啶。胸腺嘧啶也稱為5-甲基尿嘧啶,是一種嘧啶核鹼基。顧名思義,胸腺嘧啶可以透過在第 5 個碳上甲基化尿嘧啶而獲得。在RNA中,胸腺嘧啶在大多數情況下被尿嘧啶取代。在DNA中,胸腺嘧啶 (T) 透過兩個氫鍵與腺嘌呤 (A) 結合,從而穩定核酸結構。
尿嘧啶存在於RNA中,它與腺嘌呤配對,並在DNA轉錄過程中取代胸腺嘧啶。尿嘧啶的甲基化會產生胸腺嘧啶。它變成胸腺嘧啶是為了保護DNA並提高DNA複製效率。尿嘧啶可以與任何鹼基配對,具體取決於它在螺旋上的排列方式,但很容易與腺嘌呤配對,因為甲基被排斥到固定位置。尿嘧啶透過氫鍵與腺嘌呤配對。尿嘧啶是氫鍵受體,可以形成兩個氫鍵。尿嘧啶也可以與核糖結合形成核糖核苷尿苷。當磷酸基團連線到尿苷時,就會產生尿苷 5'-單磷酸。
| 含氮鹼基 | 核苷 | 脫氧核苷 |
|---|---|---|
 w:腺嘌呤 |
 w:腺苷 A |
 w:脫氧腺苷 dA |
 w:鳥嘌呤 |
 w:鳥苷 G |
 w:脫氧鳥苷 dG |
 w:胸腺嘧啶 |
 w:5-甲基尿苷 m5U |
 w:胸腺嘧啶核苷 dT |
 w:尿嘧啶 |
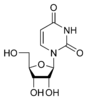 w:尿苷 U |
 w:脫氧尿苷 dU |
 w:胞嘧啶 |
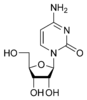 w:胞苷 C |
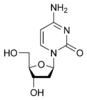 w:脫氧胞苷 dC |
核苷是糖基胺,由一個核鹼基(通常簡稱為鹼基)透過β-糖苷鍵連線到一個核糖或脫氧核糖糖上。核苷的例子包括胞苷、尿苷、腺苷、鳥苷、胸腺嘧啶核苷和肌苷。核苷可以在細胞中被特定的激酶磷酸化,在糖的伯醇基(-CH2-OH)上產生核苷酸,核苷酸是 DNA 和 RNA 的分子構建塊[13]。
核苷可以透過從頭合成途徑產生,特別是在肝臟中,但它們更多地是透過攝入和消化飲食中的核酸而提供的,其中核苷酸酶將核苷酸(如胸腺嘧啶核苷酸)分解成核苷(如胸腺嘧啶核苷)和磷酸。
1. 腺苷是一種核苷,由一個腺嘌呤分子透過β-N9-糖苷鍵連線到一個核糖糖分子(核糖呋喃糖)部分。
2. 胞苷是一種核苷分子,當胞嘧啶透過β-N1-糖苷鍵連線到一個核糖環(也稱為核糖呋喃糖)時形成。胞苷是 RNA 的組成部分。
3. 鳥苷是一種嘌呤核苷,由鳥嘌呤透過β-N9-糖苷鍵連線到一個核糖(核糖呋喃糖)環上。鳥苷可以磷酸化成為單磷酸鳥苷 (GMP)、環狀單磷酸鳥苷 (cGMP)、二磷酸鳥苷 (GDP) 和三磷酸鳥苷 (GTP)。
4. 胸腺嘧啶核苷(更準確地說稱為脫氧胸腺嘧啶核苷;也可以標記為脫氧核糖基胸腺嘧啶和胸腺嘧啶脫氧核苷)是一種化學化合物,更準確地說是一種嘧啶脫氧核苷。脫氧胸腺嘧啶核苷是 DNA 核苷 T,它在雙鏈 DNA 中與脫氧腺苷 (A) 配對。
如果胞嘧啶連線到一個脫氧核糖環上,它被稱為脫氧胞苷。[14]

一個核苷酸由一個核鹼基(含氮鹼基)、一個五碳糖(核糖或 2'-脫氧核糖)和一個到三個磷酸基組成。核鹼基和糖一起構成了一個核苷。磷酸基團與糖的 2、3 或 5 碳形成鍵,其中 5 碳位點最常見。當磷酸基團與糖的兩個羥基形成鍵時,就會形成環狀核苷酸。核糖核苷酸是糖為核糖的核苷酸,脫氧核糖核苷酸包含糖脫氧核糖。核苷酸可以包含嘌呤或嘧啶鹼基。核酸是由核苷酸單體組成的聚合的大分子。在 DNA 中,嘌呤鹼基是腺嘌呤和鳥嘌呤,而嘧啶鹼基是胸腺嘧啶和胞嘧啶。RNA 使用尿嘧啶代替胸腺嘧啶。腺嘌呤總是透過 2 個氫鍵與胸腺嘧啶配對,而鳥嘌呤透過 3 個氫鍵與胞嘧啶配對,每個配對都由於它們的獨特結構。[15]
脫氧核糖核苷酸是 DNA 或脫氧核糖核酸的單體或單個單元。每個脫氧核糖核苷酸包含三個部分:一個含氮鹼基、一個脫氧核糖糖和一個或多個磷酸基團。含氮鹼基總是與脫氧核糖的 1' 碳鍵合,脫氧核糖的 2' 碳上存在一個質子而不是 -OH 基團,這與核糖不同。磷酸基團與糖的 5' 碳鍵合。當脫氧核糖核苷酸聚合形成 DNA 時,一個核苷酸的磷酸基團將與另一個核苷酸的 3' 碳鍵合,透過脫水合成形成磷酸二酯鍵。新核苷酸總是新增到最後一個核苷酸的 3' 碳上,因此合成總是從 5' 到 3'[16]。
磷酸二酯鍵
磷酸二酯鍵是一組在磷酸基團和兩個 5 碳環碳水化合物(戊糖)之間透過兩個酯鍵形成的強共價鍵。磷酸二酯鍵對地球上大多數生命至關重要,因為它們構成了 DNA 鏈的骨架。在 DNA 和 RNA 中,磷酸二酯鍵是連線一個糖分子的 3' 碳原子和另一個糖分子的 5' 碳原子的連線,DNA 中是脫氧核糖,RNA 中是核糖。磷酸二酯鍵中的磷酸基團帶負電荷。由於磷酸基團的 pKa 接近 0,因此它們在 pH 7 時帶負電荷。這種排斥力迫使磷酸鹽佔據 DNA 鏈的相反側,並透過蛋白質(組蛋白)、金屬離子(如鎂)和多胺來中和。為了形成磷酸二酯鍵並連線核苷酸,需要將核苷酸構建塊的三磷酸或二磷酸形式分解以釋放催化酶催化反應所需的能量。當單個磷酸或兩個稱為焦磷酸的磷酸分解並催化反應時,就會形成磷酸二酯鍵。磷酸二酯鍵的水解可以被磷酸二酯酶的作用催化,磷酸二酯酶在修復 DNA 序列中起著重要作用。在生物系統中,兩個核糖核苷酸之間的磷酸二酯鍵可以被鹼性水解斷裂,因為存在遊離的 2' 羥基[17]。

 w:單磷酸腺苷 AMP |
 w:二磷酸腺苷 ADP |
 w:三磷酸腺苷 ATP |
 w:單磷酸鳥苷 GMP |
 w:二磷酸鳥苷 GDP |
 w:三磷酸鳥苷 GTP |
 w:單磷酸核糖胸腺嘧啶 rTMP |
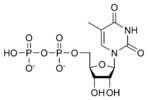 w:二磷酸核糖胸腺嘧啶 rTDP |
 w:三磷酸核糖胸腺嘧啶 rTTP |
 w:單磷酸尿苷 UMP |
 w:二磷酸尿苷 UDP |
 w:三磷酸尿苷 UTP |
 w:單磷酸胞苷 CMP |
 w:二磷酸胞苷 CDP |
 w:三磷酸胞苷 CTP |
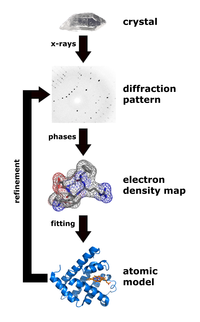
在使用標準生化技術分離和純化 DNA 後,人們會在一個罐子裡得到一個樣本,就像本文頂部的圖中一樣。以下是使用分子模型,結合晶體學和 X 射線圖案的數學分析,從定向 DNA 纖維的 X 射線衍射研究中生成結構資訊的主要步驟,這些纖維是從水合 DNA 樣本中提取的。

近晶格或近晶體是一種分子或原子晶格,其中分子排列存在相當數量(例如,大於百分之幾)的部分無序。近晶體模型的極限情況是奈米結構,例如玻璃、液體等,它們可能只有區域性有序,沒有全域性有序。以下圖顯示了二氧化矽玻璃的近晶格模型的簡單示例。
液晶也具有近晶體結構而不是晶體結構。
高水化的 B-DNA 天然存在於活細胞中,處於一種準晶態,儘管相對剛性的 DNA 雙螺旋結構透過兩條互補的螺旋 DNA 鏈中核苷酸鹼基對之間的平行氫鍵穩定(見圖),但它仍是一個動態狀態。為簡單起見,大多數 DNA 分子模型省略了動態結合到 B-DNA 的水和離子,因此對理解 B-DNA 在體內動態行為不太有用。因此,準晶態 B-DNA 的 X 射線[19][20]和光譜資料的物理和數學分析比晶態 A-DNA 的 X 射線衍射圖複雜得多。準晶模型對於 DNA 技術應用也很重要,例如DNA 奈米技術。現在,也正在開發將 DNA 的 X 射線衍射與水化活細胞的 X 射線顯微鏡相結合的新技術。[21]
A-DNA:A-DNA 是 DNA 的許多可能雙螺旋結構之一。A-DNA 被認為是三種具有生物活性的雙螺旋結構之一,另外兩種是 B-DNA 和 Z-DNA。它是一種右手雙螺旋,與更常見且眾所周知的 B-DNA 形式非常相似,但具有更短、更緊湊的螺旋結構。它似乎只出現在脫水的 DNA 樣本中,例如用於晶體學實驗的樣本,並且可能也被 DNA-RNA 雜交螺旋和雙鏈 RNA 的某些區域所採用[22]。
B-DNADNA 最常見的形式是 B-DNA。DNA 雙螺旋是核酸的螺旋聚合物,由核苷酸連線在一起,核苷酸彼此配對。在 B-DNA 中,最常見的雙螺旋結構,雙螺旋是右手螺旋,每圈大約有 10-10.5 個核苷酸。DNA 的雙螺旋結構包含一個主溝和一個次溝,主溝比次溝更寬。鑑於主溝和次溝寬度的差異,許多與 DNA 結合的蛋白質透過較寬的主溝結合。鹼基或鹼基對步的幾何形狀可以用 6 個座標來表徵:位移、滑動、上升、傾斜、滾動和扭曲。這些值精確地定義了核酸分子中每個鹼基或鹼基對相對於其沿螺旋軸的前體的空間位置和方向。它們共同表徵了分子的螺旋結構。在 DNA 或 RNA 的結構發生“正常”結構破壞的區域,這些值的改變可用於描述這種破壞。對於每個鹼基對,相對於其前體,有以下鹼基對幾何形狀需要考慮
剪下:鹼基看起來像是在 xy 平面上彼此移動。
拉伸:兩個鹼基在水平方向上被拉開。
交錯:鹼基在 xy 平面上既彼此移動又拉開。
彎曲:不接觸的鹼基末端向 z 軸向上傾斜。
螺旋扭曲:一個鹼基相對於同一鹼基對中的另一個鹼基的旋轉。
開啟:鹼基在一側接觸,但在另一側彼此傾斜。
位移:沿鹼基對平面中垂直於第一個的軸的位移,從次溝指向主溝。
滑動:沿鹼基對平面中從一條鏈指向另一條鏈的軸的位移。
上升:沿螺旋軸的位移。
傾斜:繞此軸的旋轉。
滾動:繞此軸的旋轉。
扭曲:繞螺旋軸的旋轉。
x 位移
y 位移
傾斜度
螺距:每圈螺旋的鹼基對數量。

上升和扭曲決定了螺旋的旋向和螺距。相比之下,其他座標可以為零。滑動和位移在 B-DNA 中通常很小,但在 A-DNA 和 Z-DNA 中很大。滾動和傾斜使連續的鹼基對不太平行,並且通常很小。這些座標的圖可以在 3DNA 網站上找到。請注意,“傾斜”在科學文獻中經常被不同地使用,指的是第一個鏈間鹼基對軸與螺旋軸垂直度的偏差。這對應於連續鹼基對之間的滑動,在基於螺旋的座標中,它被正確地稱為“傾斜度”。
TA-DNA:TA-DNA 是一種 DNA 形式,在結構方面與 A-DNA 最密切相關。TA-DNA 螺旋是右手螺旋,與 A-DNA 一樣,並且它兩種形式之間最明顯的區別是 TA-DNA 的鹼基對傾斜度更大,相對於螺旋軸的角度約為 50 度。因此,TA-DNA 有時被稱為傾斜的 A-DNA。[23]
Z-DNA:Z-DNA 是 DNA 的許多可能雙螺旋結構之一。它是一種左手雙螺旋結構,其中雙螺旋以之字形模式向左旋轉(而不是像更常見的 B-DNA 形式那樣向右旋轉)。Z-DNA 被認為是三種具有生物活性的雙螺旋結構之一,另外兩種是 A-DNA 和 B-DNA。Z-DNA 與右手形式完全不同。實際上,Z-DNA 通常與 B-DNA 進行比較,以說明主要差異。Z-DNA 螺旋是左手螺旋,並且具有每 2 個鹼基對重複一次的結構。與 A-DNA 和 B-DNA 不同,主溝和次溝的寬度差異很小。這種結構的形成通常是不利的,儘管某些條件可以促進它;例如交替的嘌呤-嘧啶序列(尤其是 poly(dGC)2)、負 DNA 超螺旋或高鹽和一些陽離子(所有這些都在生理溫度 37°C 和 pH 7.3-7.4 時)。Z-DNA 可以與 B-DNA 形成一個連線(稱為“B-to-Z 連線盒”),該結構涉及鹼基對的擠出。Z-DNA 構象很難研究,因為它並不作為雙螺旋的穩定特徵存在。相反,它是一種瞬態結構,偶爾由生物活性誘導,然後迅速消失。[24][25]

| A-DNA | B-DNA | Z-DNA | |
|---|---|---|---|
| 螺旋方向 | 右手 | 右手 | 左手 |
| 直徑 | 23 Å (2.3 nm) | 20 Å (2.0 nm) | 18 Å (1.8 nm) |
| 重複單元 | 1 bp | 1 bp | 2 bp |
| 旋轉/bp | 32.7° | 35.9° | 60°/2 |
| bp/圈 | 11 | 10.5 | 12 |
| bp 相對於軸的傾斜度 | +19° | −1.2° | −9° |
| 沿軸的上升/bp | 2.3 Å (0.23 nm) | 3.32 Å (0.332 nm) | 3.8 Å (0.38 nm) |
| 螺旋的螺距/圈 | 28.2 Å (2.82 nm) | 33.2 Å (3.32 nm) | 45.6 Å (4.56 nm) |
| 平均螺旋扭曲 | +18° | +16° | 0° |
| 糖苷鍵角 | 反式 | 反式 | C:反式, G:順式 |
| 糖的構象 | C3'-內式 | C2'-內式 | C:C2'-內式, G:C2'-外式 |
bp-鹼基對,nm-奈米米
DNA 的次溝和主溝:DNA 的次溝和主溝是其所屬的 DNA 型別的特徵。它們非常重要,因為它們不僅構成了 A-DNA、B-DNA 和 Z-DNA 的特徵結構,而且它們的差異決定了這些 DNA 與各種蛋白質的相互作用。這些溝中的寬度、狹窄度、深度、淺度以及靜電勢都對決定 DNA 如何與某些蛋白質反應、它將與哪些蛋白質反應以及某些部位是否可能根本不反應起著重要作用。[26]
| 次溝 | 主溝 | |
|---|---|---|
| A-DNA | 寬而淺,靜電勢相對較低 | 窄而深,靜電勢相對較高 |
| B-DNA | 窄而深,靜電勢相對較高 | 寬而淺,靜電勢相對較低 |
| Z-DNA | 窄而深,主鏈呈之字形彎曲 | 不清晰,鹼基邊緣形成凸面 |

A-DNA、B-DNA 和 Z-DNA 相對電荷的差異:對於所有形式的 DNA,整體結構都帶負電荷,整體靜電勢為負。這主要是由於 DNA 的磷酸基團,由於它們帶負電的氧原子,它們帶有一個負電荷。然而,DNA 在其外部結構上既有正電荷又有負電荷。DNA 的整體結構上的電荷分散在 A-DNA、B-DNA 和 Z-DNA 之間有所不同。A-DNA 在整個結構中大多具有負靜電勢,但主要集中在主溝部位。除了主溝之外,它還在其其餘結構中廣泛地分佈著中性靜電勢。最後,A-DNA 在其表面散佈著稀疏的正電荷。B-DNA 在其整個外部結構中大多具有負靜電勢,主要集中在主溝和次溝中。它在其表面上稀疏地散佈著一些帶正電荷的表面積,並且主要分佈在寬闊的主溝內。Z-DNA 的次溝帶負電荷,主溝通常所在的模糊區域具有輕微的正電荷部位。總體而言,Z-DNA 在其整個結構中都帶負電荷。A-DNA、B-DNA 和 Z-DNA 的結構帶負電荷。[27]
| A-DNA | B-DNA | Z-DNA | |
|---|---|---|---|
| 負勢 | 在整個結構中大量存在,但主要集中在主溝 | 在整個結構中大量存在,並在主溝和次溝中均勻集中 | 在整個全域性結構中廣泛存在,並在小溝中含量最高 |
| 正電勢 | 非常稀疏地散佈在小溝中,幾乎不在大溝中 | 非常稀疏地散佈在整個全域性結構中,主要在大溝中 | 比 A-DNA 或 B-DNA 更稀疏地分散,但位於小溝之外 |
| 中性電勢 | 在整個結構中中等程度地存在,主要位於小溝中,但不在大溝中 | 在整個結構中中等程度地分散,但含量低於 A-DNA | 在整個全域性結構中中等程度地分散,除了小溝之外 |

DNA 的螺旋結構
[edit | edit source]

DNA 超螺旋對於所有細胞中 DNA 的包裝至關重要。由於 DNA 的長度可以是細胞長度的數千倍,因此將這種遺傳物質包裝到細胞或細胞核中(真核生物)是一項艱鉅的任務。DNA 的超螺旋減少了空間,並允許包裝更多的 DNA。在原核生物中,由於環狀染色體和相對較少的遺傳物質,主要以紐結狀超螺旋為主。在真核生物中,DNA 超螺旋存在於紐結狀和螺旋狀超螺旋的多個層面上,螺旋狀超螺旋在壓縮 DNA 方面最有效。螺旋狀超螺旋透過組蛋白來實現,形成 10 奈米纖維。該纖維進一步盤繞成 30 奈米纖維,並在自身上進一步盤繞多次。在核分裂事件(如有絲分裂或減數分裂)期間,DNA 包裝會大大增加,此時 DNA 必須被壓縮並分離到子細胞中。凝聚素和粘連蛋白是染色體結構維持蛋白,有助於姐妹染色單體的凝聚和姐妹染色單體著絲粒的連線。這些 SMC 蛋白誘導正超螺旋。DNA/RNA 合成也需要超螺旋。由於 DNA 必須解開以進行 DNA/RNA 聚合酶作用,因此將產生超螺旋。聚合酶複合體前面的區域將被解開;這種應力將透過複合體前面的正超螺旋來補償。在複合體後面,DNA 被重新盤繞,並且將存在補償性負超螺旋。重要的是要注意,拓撲異構酶(如 DNA 旋轉酶(II 型拓撲異構酶))在 DNA/RNA 合成過程中緩解部分應力方面發揮著作用[28]。
NA 超螺旋可以透過連線數的變化來進行數值描述。連線數' Lk。連線數是超螺旋 DNA 最具描述性的性質。Lko,鬆弛(B 型)DNA 質粒/分子的匝數,透過將分子的總鹼基對除以鬆弛的 bp/匝數來確定,具體取決於參考文獻,為 10.4-10.5。
Lk 僅僅是指單鏈在平面投影中穿過另一鏈的次數。DNA 的拓撲結構由下面的方程描述,其中連線數等效於 TW 的總和,TW 是雙螺旋的匝數或圈數,而 Wr 是卷數或“纏繞度”。如果存在一個閉合的 DNA 分子,則 TW 和 Wr 的總和,或連線數,不會改變。但是,TW 和 Wr 可以發生互補變化,而不會改變它們的總和。
連線數的變化,ΔLk,是質粒/分子中的實際匝數 Lk 減去鬆弛質粒/分子 Lko 中的匝數。
如果 DNA 呈負超螺旋,則 ΔLk < 0。負超螺旋意味著 DNA 被欠纏繞。
與分子大小無關的標準表達方式是“特定連線差異”或“超螺旋密度”,用 σ 表示。σ 表示相對於鬆弛分子/質粒中的總匝數新增或去除的匝數,指示超螺旋的程度。
連線數是一個數值不變數,它描述了三維空間中兩條閉合曲線的連線。直觀地說,連線數代表每條曲線繞另一條曲線纏繞的次數。連線數始終為整數,但根據兩條曲線的方向可以為正或負[31]。由於超螺旋 DNA 的連線數 *L* 是兩條鏈相互交織的次數(並且兩條鏈都保持共價完整),*L* 不能改變。環狀 DNA 雙鏈體的參考狀態(或引數)*L0* 是其鬆弛狀態。在這種狀態下,其纏繞度 *W* = 0。由於 *L = T + W*,在鬆弛狀態下 *T = L*。因此,如果我們有一個 400 bp 的鬆弛環狀 DNA 雙鏈體,則 *L ~ 40*(假設 B-DNA 每圈 ~10 bp)。那麼 *T ~ 40*。
- 正超螺旋
- T = 0,W = 0,則 L = 0
- T = +3,W = 0,則 L = +3
- T = +2,W = +1,則 L = +3
- 負超螺旋
- T = 0,W = 0,則 L = 0
- T = -3,W = 0,則 L = -3
- T = -2,W = -1,則 L = -3
負超螺旋有利於 DNA 的區域性解開,從而允許進行諸如轉錄、DNA 複製和重組等過程。負超螺旋也被認為有利於 B-DNA 和Z-DNA之間的轉變,並調節參與基因調控的 DNA 結合蛋白的相互作用。[32][33]
DNA 測序
[edit | edit source]RNA測序是最早的核苷酸測序方法之一。 RNA測序的一個重要里程碑是第一個完整基因和噬菌體MS2的完整基因組的測序,由Walter Fiers及其在根特大學(比利時根特)的同事在1972年至1976年間鑑定並發表。 在20世紀70年代初,Frederick Sanger在英國劍橋大學,以及Walter Gilbert和Allan Maxam在哈佛大學開發出快速DNA測序方法之前,人們採用了許多費力的方法。 例如,1973年,Gilbert和Maxam使用一種稱為“遊點分析”的方法報道了24個鹼基對的序列。 Sanger及其同事在1975年開發的鏈終止法很快成為首選方法,因為它相對簡單且可靠。 [34]
Maxam和Gilbert方法
[edit | edit source]1976年至1977年,Allan Maxam和Walter Gilbert開發了一種DNA測序方法,該方法基於DNA的化學修飾以及隨後在特定鹼基處的裂解。 儘管Maxam和Gilbert在Sanger和Coulson關於“正負”測序的開創性論文發表兩年後才發表了他們的化學測序方法,但Maxam-Gilbert測序迅速變得更加流行,因為可以直接使用純化的DNA,而最初的Sanger方法則要求每個讀取起點都被克隆用於生產單鏈DNA。 然而,隨著鏈終止法的改進(見下文),Maxam-Gilbert測序已經不受歡迎,因為它的技術複雜性使其無法在標準分子生物學試劑盒中使用,需要大量使用有害化學品,並且難以放大。 該方法需要在DNA的一個5'末端進行放射性標記(通常透過使用γ-32P ATP的激酶反應),並純化待測序的DNA片段。 化學處理會在四個反應中的一個或兩個中對四個核苷酸鹼基中的很小一部分產生斷裂(G、A+G、C、C+T)。 例如,嘌呤(A+G)使用甲酸進行脫嘌呤,鳥嘌呤(以及一定程度上的腺嘌呤)使用二甲基硫酸進行甲基化,嘧啶(C+T)使用聯氨進行甲基化。 在聯氨反應中新增鹽(氯化鈉)可以抑制胸腺嘧啶在僅C反應中的甲基化。 然後,修飾的DNA在修飾鹼基的位置用熱哌啶裂解。 修飾化學品的濃度控制在平均每個DNA分子引入一個修飾。 因此,產生了一系列標記片段,從放射性標記的末端到每個分子中第一個“切割”位點。 在變性丙烯醯胺凝膠中並排電泳四個反應中的片段,以進行尺寸分離。 為了視覺化片段,將凝膠暴露於X射線片上進行放射自顯影,產生一系列暗帶,每個暗帶對應於一個放射性標記的DNA片段,由此可以推斷出序列。 該方法也稱為“化學測序”,它導致了用於繪製DNA結合蛋白的DNA結合位點的甲基化干擾分析法。 [35]
雙脫氧核苷酸鏈終止法
[edit | edit source]
由於鏈終止法(或以其開發者 Frederick Sanger 命名的 Sanger 方法)比 Maxam 和 Gilbert 的方法更有效,並且使用的有毒化學物質和放射性物質更少,因此它迅速成為首選方法。 Sanger 方法的關鍵原理是使用雙脫氧核苷酸三磷酸 (ddNTPs) 作為 DNA 鏈終止劑。
經典的鏈終止法需要單鏈 DNA 模板、DNA 引物、DNA 聚合酶、正常的脫氧核苷酸磷酸 (dNTPs) 和修飾的核苷酸 (雙脫氧核苷酸)。 這些 ddNTPs 也會被放射性標記或熒游標記,以便在自動測序儀中檢測。 將 DNA 樣品分成四個獨立的測序反應,每個反應包含所有四種標準脫氧核苷酸 (dATP、dGTP、dCTP 和 dTTP) 以及 DNA 聚合酶。 在每個反應中只新增四種雙脫氧核苷酸中的一種 (ddATP、ddGTP、ddCTP 或 ddTTP),它們是鏈終止核苷酸,缺少形成兩個核苷酸之間磷酸二酯鍵所需的 3'-羥基 (OH) 基團,因此終止了 DNA 鏈的延伸,並導致了不同長度的 DNA 片段。 [36]
新合成的標記 DNA 片段被熱變性,並根據尺寸(解析度僅為一個核苷酸)透過變性聚丙烯醯胺-尿素凝膠電泳分離,四個反應中的每一個都在四個獨立的泳道 (泳道 A、T、G、C) 中執行;然後透過放射自顯影或紫外光視覺化 DNA 帶,並且可以從 X 射線片或凝膠影像直接讀取 DNA 序列。 在右側的影像中,將 X 射線片暴露於凝膠中,暗帶對應於不同長度的 DNA 片段。 泳道中的暗帶表示在摻入雙脫氧核苷酸 (ddATP、ddGTP、ddCTP 或 ddTTP) 後鏈終止導致的 DNA 片段。 然後使用四個泳道中不同帶的相對位置從下到上讀取 DNA 序列。 [37]

鏈終止測序的技術變體包括使用含有放射性磷的核苷酸進行放射性標記,或使用在 5' 端用熒光染料標記的引物。 染料引物測序有利於在光學系統中讀取,以便更快、更經濟地分析和自動化。 由 Leroy Hood 及其同事 [38][39] 開發的熒游標記 ddNTPs 和引物為自動化的、高通量的 DNA 測序奠定了基礎。

鏈終止法極大地簡化了 DNA 測序。 例如,市面上有鏈終止法試劑盒,其中包含測序所需的試劑,已經預先分裝好,可以隨時使用。 侷限性包括引物與 DNA 的非特異性結合,影響 DNA 序列的準確讀取,以及 DNA 二級結構影響序列的保真度。 [40]
染料終止子測序
[edit | edit source]
染料終止子測序 使用鏈終止 ddNTPs 的標記,允許在一個反應中進行測序,而不是像標記引物法那樣進行四個反應。 在染料終止子測序中,四種雙脫氧核苷酸鏈終止劑中的每一種都用熒光染料標記,每種染料在不同的波長髮射光。
由於其更高的效率和速度,染料終止子測序現在是自動化測序的主流方法。 其侷限性包括由於染料標記的鏈終止劑摻入 DNA 片段的差異而導致的染料效應,導致在 色譜圖 中的電子 DNA 序列軌跡出現峰高和峰形不均。 毛細管電泳(見左側圖)。
這個問題已經透過使用改進的 DNA 聚合酶酶系統和最小化摻入變異性的染料以及消除“染料斑點”的方法得到解決。 染料終止子測序方法以及自動化的、高通量的 DNA 序列分析儀現在正被用於絕大多數測序專案。 [41]
挑戰
[edit | edit source]DNA 測序的常見挑戰包括序列前 15-40 個鹼基的質量較差,以及在 700-900 個鹼基後的測序軌跡質量下降。 鹼基識別 軟體通常會給出質量估計,以幫助進行質量修剪。 [42][43]
在 DNA 片段在測序之前被克隆的情況下,所得序列可能包含克隆載體的部分。 相反,基於 PCR 的克隆和新興的基於焦磷酸測序的測序技術通常避免使用克隆載體。 最近,開發出一種步法 Sanger 測序(結合擴增和測序)方法,例如 Ampliseq 和 SeqSharp,它們允許在不克隆或事先擴增的情況下快速測序目標基因。 [44][45][46]
當前方法只能在一個反應中直接測序相對較短的(300-1000 核苷酸 長)DNA 片段。 測序大於此尺寸限制的 DNA 片段的主要障礙是缺乏區分僅在長度上相差一個核苷酸的大型 DNA 片段的分離能力。 在所有情況下,使用具有遊離 5' 末端的引物是必不可少的。 [47]
自動化和樣本製備
[edit | edit source]
自動 DNA 測序儀 (DNA 測序儀) 可以在一次批次 (執行) 中對多達 384 個 DNA 樣本進行測序,每天最多可以執行 24 次。DNA 測序儀進行毛細管電泳以進行大小分離、檢測和記錄染料熒光,並將資料輸出為熒光峰跡色譜圖。測序反應透過熱迴圈、清理和重新懸浮在緩衝溶液中,然後裝載到測序儀上,這些步驟是單獨執行的。許多商業和非商業軟體包可以自動修剪低質量的 DNA 跡線。這些程式會評估每個峰的質量並去除低質量的鹼基峰(通常位於序列的末端)。此類演算法的準確性低於人工操作員的目視檢查,但足以自動處理大型序列資料集[48]。
- 1869 年,瑞士醫生弗里德里希·米歇爾從丟棄的繃帶膿液中首次分離出 DNA,他發現了一種顯微鏡下可見的物質。
- 1937 年,威廉·阿斯特伯裡製作了第一個 X 射線衍射圖譜,表明 DNA 具有規則的結構。
- 1928 年,弗雷德里克·格里菲斯發現,將殺死的“光滑型”肺炎球菌與活的“粗糙型”肺炎球菌混合,可以將“光滑型”的特徵轉移到“粗糙型”。
- 1952 年,阿爾弗雷德·赫希和瑪莎·蔡斯在赫希-蔡斯實驗中表明,DNA 是 T2噬菌體的遺傳物質。
- 1953 年,詹姆斯·D·沃森和弗朗西斯·克里克提出了 DNA 結構的雙螺旋模型。
- 1972 年,重組 DNA 技術的開發,允許分離出明確的 DNA 片段;在此之前,唯一可用於測序的樣本來自噬菌體或病毒 DNA。
- 1977 年,第一個完整的 DNA 基因組被測序,即噬菌體 φX174。
- 1977 年,艾倫·馬克薩姆和沃爾特·吉爾伯特發表了“化學降解法測序 DNA”。弗雷德里克·桑格獨立發表了“用鏈終止抑制劑測序 DNA”。
- 1984 年,英國醫學研究委員會的科學家破譯了艾伯斯坦-巴爾病毒(170 kb)的完整 DNA 序列。
- 1986 年,加州理工學院勒羅伊·E·胡德的實驗室和史密斯宣佈了第一臺半自動 DNA 測序儀。
- 1987 年,應用生物系統公司推出了第一臺自動化測序儀,型號為 ABI 370。
- 1990 年,美國國立衛生研究院 (NIH) 開始對解螺旋支原體、大腸桿菌、秀麗隱杆線蟲和釀酒酵母進行大規模測序試驗(每鹼基 0.75 美元)。
- 1991 年,克雷格·文特爾的實驗室開始對人類表達序列標籤進行測序,試圖捕獲人類基因組的編碼部分。
- 1995 年,克雷格·文特爾、漢密爾頓·史密斯及其同事在基因組研究所 (TIGR) 發表了第一個自由生活生物體的完整基因組,即嗜血桿菌。這個環狀染色體包含 1,830,137 個鹼基,其發表在《科學》雜誌上標誌著首次使用全基因組鳥槍法測序,消除了對初始作圖工作的需求。
- 1996 年,帕爾·尼倫和他的學生穆斯塔法·羅納吉在斯德哥爾摩的皇家理工學院發表了他們的焦磷酸測序方法。
- 1998 年,華盛頓大學的菲爾·格林和布倫特·尤因發表了用於測序資料分析的“phred”。
- 2000 年,Lynx Therapeutics 釋出並推出了“MPSS”——一種並行、介面卡/連線介導的、基於珠子的測序技術,開啟了“下一代”測序。
- 2001 年,人類基因組草圖發表。
- 2004 年,454 Life Sciences 推出了一種並行的焦磷酸測序版本。他們機器的第一版將測序成本降低了 6 倍,與自動桑格測序相比,這是繼 MPSS 之後新一代測序技術中的第二種。
- ↑ http://en.wikipedia.org/w/index.php?title=DNA&oldid=424928563
- ↑ Berry, Andrew; Watson, James D. (2003). DNA: the secret of life. New York: Alfred A. Knopf. ISBN 0-375-41546-7.
- ↑ http://en.wikipedia.org/w/index.php?title=DNA&oldid=424928563
- ↑ Berry, Andrew; Watson, James D. (2003). DNA: the secret of life. New York: Alfred A. Knopf. ISBN 0-375-41546-7.
- ↑ 格里菲斯實驗
- ↑ 赫希-蔡斯實驗
- ↑ Hershey, A.D. and Chase, M. (1952) Independent functions of viral protein and nucleic acid in growth of bacteriophage. J Gen Physiol. 36:39–56.
- ↑ 艾弗裡-麥克勞德-麥卡錫實驗
- ↑ http://en.wikipedia.org/w/index.php?title=DNA&oldid=427633053
- ↑ 鹼基對
- ↑ 嘧啶
- ↑ 胞嘧啶
- ↑ http://en.wikipedia.org/w/index.php?title=Nucleoside&oldid=419856059
- ↑ http://en.wikipedia.org/w/index.php?title=Nucleoside&oldid=419856059
- ↑ http://en.wikipedia.org/w/index.php?title=Nucleotide&oldid=422383586
- ↑ http://en.wikipedia.org/w/index.php?title=Nucleotide&oldid=422383586
- ↑ http://en.wikipedia.org/w/index.php?title=Phosphodiester_bond&oldid=407638927
- ↑ Herbert R. Wilson, FRS. Diffraction of X-rays by proteins, Nucleic Acids and Viruses., London: Edward Arnold (Publishers) Ltd. 1966.
- ↑ Hosemann R., Bagchi R.N., Direct analysis of diffraction by matter, North-Holland Publs., Amsterdam – New York, 1962.
- ↑ Baianu, I.C. (1978). "X-ray scattering by partially disordered membrane systems". Acta Cryst.,. A34 (5): 751–3. doi:10.1107/S0567739478001540.
{{cite journal}}: CS1 maint: extra punctuation (link) - ↑ 山本 陽,篠原 功. (2002). "X 射線顯微鏡在活體水化細胞分析中的應用". 解剖學記錄. 269 (5): 217–23. doi:10.1002/ar.10166. PMID 12379938.
{{cite journal}}: 未知引數|month=被忽略 (幫助) - ↑ http://en.wikipedia.org/w/index.php?title=A-DNA&oldid=379025458
- ↑ Rohs, Remo, Xiangshu Jin, Sean M. West, Rohit Joshi, Barry Honig, 和 Richard S. Mann. "蛋白質-DNA 識別特異性的起源." 年度評論. 無名氏, 24 03 2010. 網路. 2011 年 10 月 29 日. <http://www.annualreviews.org/doi/full/10.1146/annurev-biochem-060408-091030?url_ver=Z39.88-2003&rfr_id=ori:rid:crossref.org&rfr_dat=cr_pub=pubmed>.
- ↑ http://en.wikipedia.org/w/index.php?title=Z-DNA&oldid=418927717
- ↑ Zhang H, Yu H, Ren J, Qu X. (2006). "立方烷狀銪-L-天冬氨酸複合物在低鹽條件下的可逆B/Z-DNA轉變和非B型polydApolydT選擇性". 生物物理學雜誌. 90 (9): 3203–3207.
- ↑ Rohs, Remo, Xiangshu Jin, Sean M. West, Rohit Joshi, Barry Honig, 和 Richard S. Mann. "蛋白質-DNA 識別特異性的起源." 年度評論. 無名氏, 24 03 2010. 網路. 2011 年 10 月 29 日. <http://www.annualreviews.org/doi/full/10.1146/annurev-biochem-060408-091030?url_ver=Z39.88-2003&rfr_id=ori:rid:crossref.org&rfr_dat=cr_pub=pubmed>.
- ↑ Rohs, Remo, Xiangshu Jin, Sean M. West, Rohit Joshi, Barry Honig, 和 Richard S. Mann. "蛋白質-DNA 識別特異性的起源." 年度評論. 無名氏, 24 03 2010. 網路. 2011 年 10 月 29 日. <http://www.annualreviews.org/doi/full/10.1146/annurev-biochem-060408-091030?url_ver=Z39.88-2003&rfr_id=ori:rid:crossref.org&rfr_dat=cr_pub=pubmed>.
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_supercoil&oldid=417575151
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_supercoil&oldid=417575151
- ↑ Vologodskii AV, Lukashin AV, Anshelevich VV 等. (1979). "超螺旋 DNA 的波動". 核酸研究. 6: 967–682. doi:10.1093/nar/6.3.967.
{{cite journal}}: 在|author=中顯式使用 et al. (幫助)CS1 maint: 多個名稱:作者列表 (連結) - ↑ http://en.wikipedia.org/w/index.php?title=DNA_supercoil&oldid=417575151
- ↑ H. S. Chawla. (2002). 植物生物技術導論. 科學出版社. ISBN 1578082285.
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_supercoil&oldid=417575151
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407
- ↑ Smith LM, Sanders JZ, Kaiser RJ 等. (1986). "自動 DNA 序列分析中的熒光檢測". 自然. 321 (6071): 674–9. doi:10.1038/321674a0. PMID 3713851.
我們已經開發出一種用於 DNA 序列分析部分自動化的方法。 DNA 片段的熒光檢測是透過共價連線到酶促 DNA 序列分析中使用的寡核苷酸引物的熒光團來實現的。 對於每個特定於鹼基 A、C、G 和 T 的反應,使用不同的彩色熒光團。 反應混合物被合併並共同電泳透過單個聚丙烯醯胺凝膠管,分離的 DNA 熒光帶在管底部附近被檢測到,序列資訊直接由計算機獲取。
{{cite journal}}: 在|author=中顯式使用 et al. (幫助)CS1 maint: 多個名稱:作者列表 (連結) - ↑ Smith LM, Fung S, Hunkapiller MW, Hunkapiller TJ, Hood LE. (1985). "在 5' 末端含有脂肪族氨基的寡核苷酸的合成:用於 DNA 序列分析的熒光 DNA 引物的合成". 核酸研究. 13 (7): 2399–412. doi:10.1093/nar/13.7.2399. PMC 341163. PMID 4000959.
{{cite journal}}: 未知引數|month=被忽略 (幫助)CS1 maint: 多個名稱:作者列表 (連結) - ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407
- ↑ "Phred - 質量鹼基呼叫". 檢索於 2011-02-24.
- ↑ "下一代測序平臺的鹼基呼叫 - 簡要生物資訊學". 檢索於 2011-02-24.
- ↑ Murphy, K.; Berg, K.; Eshleman, J. (2005). "透過組合擴增和迴圈測序反應對基因組 DNA 進行測序". 臨床化學. 51 (1): 35–39.
- ↑ Sengupta, D.; Cookson, B. (2010). "SeqSharp: 一種改進迴圈測序的通用方法,促進穩健的一步式聯合擴增和測序方法"。分子診斷雜誌:JMD 12 (3): 272–277。
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407
- ↑ http://en.wikipedia.org/w/index.php?title=DNA_sequencing&oldid=422803407