MINC/工具/mni autoreg
在診斷和研究應用中,當不同的資料集可以透過對等解剖平面的視覺檢查進行比較時,人類大腦磁共振(MR)影像的解釋會更容易。使用預定義的圖譜模板進行定量分析通常需要先將圖譜和影像平面對齊。不幸的是,在不同的掃描會話中獲得的軸向平面在它們的相對位置和方向上往往不同,並且這些切片與圖譜中的切片不共面。已經開發出一種完全自動的方法,基於多尺度三維(3D)互相關,將給定的體積資料集配準到與 Talairach 立體定向座標系對齊的平均 MRI 大腦(n > 300)。一旦資料集透過演算法恢復的變換進行重取樣,圖譜切片就可以直接疊加在重取樣體積的相應切片上(見下文)。使用這種標準化空間還允許直接比較,逐個體素地比較,兩個或多個被帶入立體定向空間的資料集。
一個 Perl 指令碼 (mritotal) 實施了多解析度擬合策略,該策略已用於將 500 多個大腦對映到蒙特利爾神經病學研究所的立體定向空間。此過程的核心是 minctracc,該程式自動找到將一個體積資料集(儲存在 MINC 格式中,見下文)對映到另一個數據集的最佳線性變換。該程式使用對使用者可選引數數量的最佳化來識別最佳(根據使用者選擇的目標函式)變換,將第一個資料集的體素值對映到第二個資料集。
配準的主要指令碼是
- mritotal - 呼叫 mincblur 和 minctracc 來實現立體定向配準的指令碼
- mritoself - 腦內配準
其他安裝的指令碼或程式是
- autocrop - 用於提取和操作 MINC 檔案的邊界
- make_model - 生成 `mritotal` 所需的適當模糊和子取樣影像
- make_phantom - 生成橢球體或矩形幻影影像
- mincbbox - 提取影像的邊界框
- mincblur - 用於使用高斯模糊核進行體積卷積的程式
- mincchamfer - 計算影像的 Chamfer 距離變換
- minctracc - 用於估計配準所需變換的程式
- xfmtool - 對變換 (.xfm) 檔案執行各種操作
其中一些附帶手冊頁,並且所有都響應 `-help`。
mritotal 需要兩個外部檔案才能執行。它們是配置檔案 (mritotal.cfg) 和協議檔案(預設值為 mritotal.default.cfg,但您可以使用 -protocol 選項指定其他檔案)。如果您遵循以上所有說明,則這些檔案將被正確自定義並安裝到您的站點。
但是,如果出現問題,您應該知道以下內容
- 安裝配置檔案和協議檔案(s) 的目錄預設設定為 /usr/local/bic/etc/mni_autoreg。
- 模型的位置在 mritotal.cfg 中給出(或在 mritotal 中使用 -modeldir 選項給出)。
- 如果您發現有必要為您的站點資料建立新的協議(例如,如果您的 MRI 資料始終涵蓋比大腦更多的內容,但預設協議使用的啟發式方法不起作用 - 有關更多資訊,請參閱 mritotal 手冊頁),那麼您應該複製並更改預設協議檔案。例如,您可能想將協議命名為“mysite” - 在這種情況下,將 mritotal.default.cfg 複製到 mritotal.mysite.cfg,並確保編輯 mritotal.cfg 以使它預設使用新的協議。也就是說,確保它具有“-protocol mysite”而不是“-protocol default”。
為了更好地理解 mni_autoreg 或線性配準是如何工作的,這裡有一個簡單的例子。假設我們有一張個人的影像,並且希望將其與模型對齊。這兩張影像顯示在下面,左邊是個人影像 (indiv.mnc),右邊是模型 (model.mnc)。請注意,這兩個檔案具有不同的取樣和體素大小,如白色線條所示
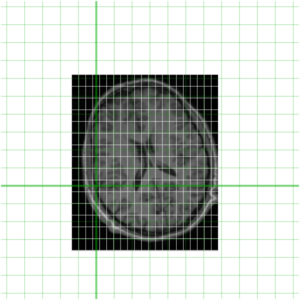
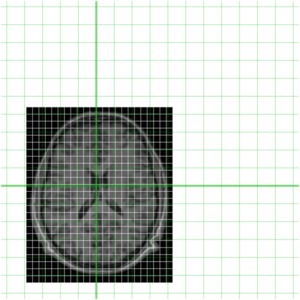
當然,作為 MINC 檔案,這兩個檔案都代表了世界空間的取樣(由起點、步長和方向餘弦定義 - 使用 mincinfo 檢視)。為了更好地理解這一點,我們的兩個檔案再次顯示在下面,它們的相對位置在世界空間中用綠色顯示。
為了瞭解它們目前是如何重疊的,我在下面將它們放在一起
很明顯,它們沒有相互對齊或“配準”,因此我們必須使用 minctracc 或 mritoself 等工具來對齊它們
為此,我們可以使用這樣的命令
minctracc -lsq9 indiv.mnc model.mnc align.xfm
這裡的 -lsq9 引數指示 minctracc 恢復 3 個旋轉、3 個平移和 3 個縮放。請注意,此步驟的輸出不是對齊的影像,而是輸出(線性)變換檔案,其中包含一個 4x4 變換矩陣。它由 3x3 仿射變換加上 3 個平移組成(如果需要的話)。下面是其中一個檔案的示例
MNI Transform File Transform_Type = Linear; Linear_Transform = 0.995668319594959 -0.30943698305448 0.0180935790540566 8.9918311534818 0.320977051206147 0.94080272211886 0.00947446085537751 -39.3894632128575 -0.0222220364958048 -0.00403786869719625 1.15379667282104 -33.4353790283203;
MNI 變換檔案有一個隱式的第 4 行,由 0,0,0,1 組成,但此資料未儲存在檔案中。為了瞭解此矩陣的分解是什麼樣的,我們可以使用 xfm2param
xfm2param align.xfm
這將生成如下輸出
after parameter extraction -center 0.00000 0.00000 0.00000 -translation 8.99183 -39.38946 -33.43538 -rotation -0.20051 1.10337 17.25793 -scale 1.04280 0.99372 1.15402 -shear 0.02764 -0.00000 -0.00000
因此,現在我們有了變換,我們需要對原始輸入資料進行重取樣。為此,我們使用 mincresample,但是我們有幾個關於輸出檔案取樣的選擇。第一個也是最常用的方法是這樣的
mincresample -like model.mnc -transformation align.xfm source.mnc resampled.mnc
請注意,輸出檔案現在具有與原始模型相同的取樣。
下一個選項是這樣的
mincresample -tfm_input_sampling -transformation align.xfm source.mnc resampled.mnc
這指示 mincresample 透過輸入變換重取樣檔案的原始取樣,請注意,這將導致一個帶有方向餘弦的檔案。
最後一個選項是使用個人的原始取樣,如下所示
mincresample -use_input_sampling -transformation align.xfm source.mnc resampled.mnc
使用這種方法將保留原始取樣,但也會保留檔案的原始位置,這意味著您可能會或可能不會保留檔案的完全“覆蓋”。
因此,-like 選項是最常用的方法,因為它允許對生成的重取樣檔案使用基於體素的工具(如 minccalc 和 mincmath)進行進一步處理
版權所有 (C) 1993-2010 Louis Collins 和 Greg Ward,麥吉爾大學蒙特利爾神經病學研究所麥康奈爾腦成像中心。特此授予您在任何目的和無償情況下使用、複製、修改和分發此軟體及其文件的許可權,但前提是在所有副本中保留上述版權宣告。作者和麥吉爾大學對該軟體的適用性不作任何陳述。它按“原樣”提供,不提供明示或暗示的保證。對於因使用或誤用此軟體包而導致的任何資料丟失、裝置損壞、財產損失或對受試者或患者的傷害,作者概不負責。所有使用 MNI_AutoReg 配準的結果都應該始終在所有三個空間維度上透過視覺檢查仔細檢查。MNI_AutoReg 配準決不能用於可能導致患者或受試者受傷或導致誤診的環境中。此包僅供研究使用。
我們不保證此包沒有概念或程式設計錯誤。提供此包給您並不意味著或保證提供超出本文件中提供的任何額外支援。同樣,它也不保證您會收到有關發現的任何程式設計錯誤的通知,或會收到軟體的未來更新版本。作者不對因您使用蒙特利爾神經病學研究所自動線性配準包而造成的直接或間接損失承擔任何責任。
我們希望至少引用以下文章來描述在使用 MNI_AutoReg 包分析資料的任何出版物中使用的配準方法
DL Collins,P Neelin,TM Peters 和 AC Evans,自動 3D 跨主體配準 MRI 體積資料到標準化 Talairach 空間,計算機輔助斷層掃描雜誌,18(2) p192-205, 1994
D. L. Collins, P. Neelin, T. M. Peters 和 A. C. Evans, ``自動 3D 跨主體配準 MRI 體積資料到標準化 Talairach 空間, 計算機輔助斷層掃描雜誌,18(2) pp192-205, 1994.